3.1. التحقق المتبادل: تقييم أداء المقدر#
يُعد تعلم معلمات دالة التنبؤ واختبارها على نفس البيانات خطأً منهجيًا: النموذج الذي يكرر فقط
تسميات العينات التي رآها للتو ستحصل على درجة مثالية لكنها ستفشل في التنبؤ بأي شيء مفيد على البيانات التي لم يتم رؤيتها بعد.
تسمى هذه الحالة الإفراط في التجهيز.
لتجنب ذلك، من الممارسات الشائعة عند إجراء
تجربة تعلم آلي (خاضعة للإشراف)
لإخفاء جزء من البيانات المتاحة كمجموعة اختبار X_test, y_test.
لاحظ أن كلمة "تجربة" لا يُقصد بها
للدلالة على الاستخدام الأكاديمي فقط،
لأنه حتى في البيئات التجارية
عادة ما يبدأ التعلم الآلي بشكل تجريبي.
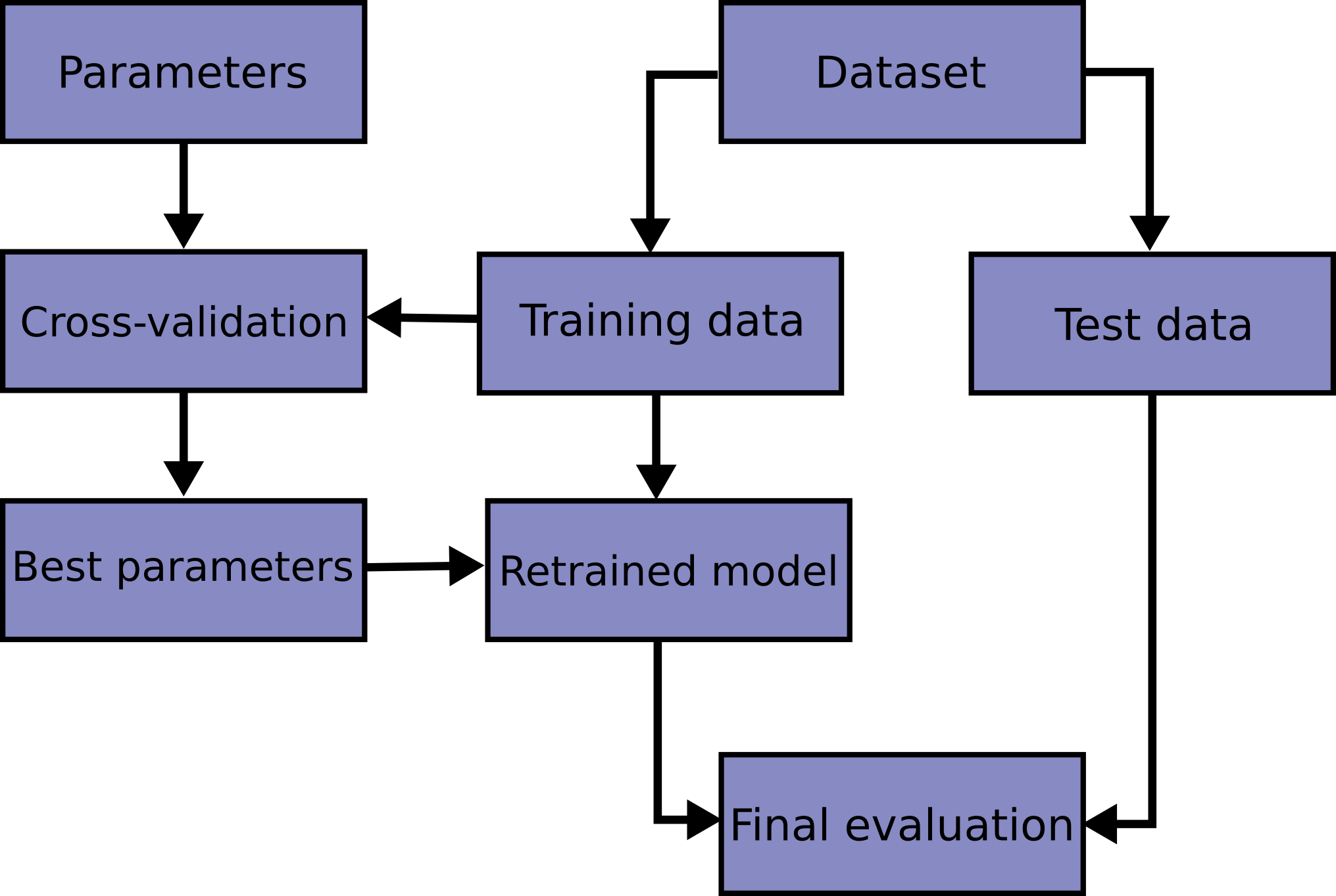
فيما يلي مخطط انسيابي لسير عمل التحقق المتبادل النموذجي في تدريب النموذج.
يمكن تحديد أفضل المعلمات بواسطة
بحث الشبكة التقنيات.
في scikit-learn، تقسيم عشوائي إلى مجموعات التدريب والاختبار
يمكن حسابها بسرعة باستخدام دالة المساعدة train_test_split.
دعنا نحمل مجموعة بيانات iris لتناسب جهاز متجه دعم خطي عليها:
>>> import numpy as np
>>> from sklearn.model_selection import train_test_split
>>> from sklearn import datasets
>>> from sklearn import svm
>>> X, y = datasets.load_iris(return_X_y=True)
>>> X.shape, y.shape
((150, 4), (150,))
يمكننا الآن أخذ عينة سريعة من مجموعة التدريب مع الاحتفاظ بـ 40٪ من البيانات لاختبار (تقييم) المصنف الخاص بنا:
>>> X_train, X_test, y_train, y_test = train_test_split(
... X, y, test_size=0.4, random_state=0)
>>> X_train.shape, y_train.shape
((90, 4), (90,))
>>> X_test.shape, y_test.shape
((60, 4), (60,))
>>> clf = svm.SVC(kernel='linear', C=1).fit(X_train, y_train)
>>> clf.score(X_test, y_test)
0.96...
عند تقييم إعدادات مختلفة ("معلمات فائقة") للمقدرات،
مثل إعداد C الذي يجب تعيينه يدويًا لـ SVM،
لا يزال هناك خطر الإفراط في التجهيز على مجموعة الاختبار
لأنه يمكن تعديل المعلمات حتى يعمل المقدر على النحو الأمثل.
بهذه الطريقة، يمكن أن "تتسرب" المعرفة حول مجموعة الاختبار إلى النموذج
ولم تعد مقاييس التقييم تُبلغ عن أداء التعميم.
لحل هذه المشكلة، يمكن الاحتفاظ بجزء آخر من مجموعة البيانات
كمجموعة تسمى "مجموعة التحقق من الصحة": يستمر التدريب على مجموعة التدريب،
بعد ذلك يتم التقييم على مجموعة التحقق من الصحة،
وعندما تبدو التجربة ناجحة،
يمكن إجراء التقييم النهائي على مجموعة الاختبار.
ومع ذلك، عن طريق تقسيم البيانات المتاحة إلى ثلاث مجموعات، نحن نقلل بشكل كبير من عدد العينات التي يمكن استخدامها لتعلم النموذج، ويمكن أن تعتمد النتائج على اختيار عشوائي معين لزوج مجموعات (التدريب، التحقق من الصحة).
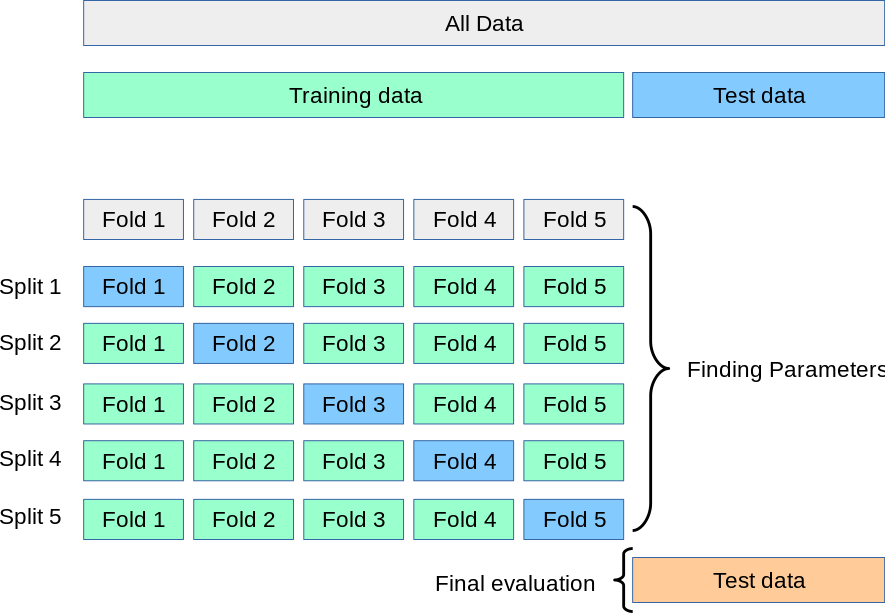
الحل لهذه المشكلة هو إجراء يسمى التحقق المتبادل (CV للاختصار). يجب أن تظل مجموعة الاختبار محجوزة للتقييم النهائي، لكن لم تعد هناك حاجة إلى مجموعة التحقق من الصحة عند القيام بـ CV. في النهج الأساسي، يسمى k-fold CV، يتم تقسيم مجموعة التدريب إلى k مجموعات أصغر (تم وصف مناهج أخرى أدناه، لكن بشكل عام اتبع نفس المبادئ). يتم اتباع الإجراء التالي لكل من "الطيات" k:
يتم تدريب نموذج باستخدام \(k-1\) من الطيات كبيانات تدريب؛
يتم التحقق من صحة النموذج الناتج على الجزء المتبقي من البيانات (على سبيل المثال، يتم استخدامه كمجموعة اختبار لحساب مقياس أداء مثل الدقة).
مقياس الأداء الذي أبلغ عنه التحقق المتبادل k-fold هو متوسط القيم المحسوبة في الحلقة. يمكن أن يكون هذا النهج مكلفًا من الناحية الحسابية، لكنها لا تهدر الكثير من البيانات (كما هو الحال عند إصلاح مجموعة تحقق من الصحة التعسفية)، وهي ميزة رئيسية في مشاكل مثل الاستدلال العكسي حيث يكون عدد العينات صغيرًا جدًا.
3.1.1. حساب المقاييس المتحقق منها#
أبسط طريقة لاستخدام التحقق المتبادل هي استدعاء
دالة المساعدة cross_val_score على المقدر ومجموعة البيانات.
يوضح المثال التالي كيفية تقدير دقة نواة خطية دعم آلة المتجهات على مجموعة بيانات iris عن طريق تقسيم البيانات، وملاءمة نموذج وحساب النتيجة 5 مرات متتالية (مع انقسامات مختلفة في كل مرة زمن):
>>> from sklearn.model_selection import cross_val_score
>>> clf = svm.SVC(kernel='linear', C=1, random_state=42)
>>> scores = cross_val_score(clf, X, y, cv=5)
>>> scores
array([0.96..., 1. , 0.96..., 0.96..., 1. ])
ومن ثم يتم إعطاء متوسط الدرجة والانحراف المعياري بواسطة:
>>> print("%0.2f accuracy with a standard deviation of %0.2f" % (scores.mean(), scores.std()))
0.98 دقة مع انحراف معياري قدره 0.02
افتراضيًا، يتم حساب النتيجة في كل تكرار CV هي النتيجة
طريقة المقدر. من الممكن تغيير هذا باستخدام
معلمة التسجيل:
>>> from sklearn import metrics
>>> scores = cross_val_score(
... clf, X, y, cv=5, scoring='f1_macro')
>>> scores
array([0.96..., 1. ..., 0.96..., 0.96..., 1. ])
انظر معلمة scoring: تعريف قواعد تقييم النموذج للحصول على التفاصيل. في حالة مجموعة بيانات Iris، يتم موازنة العينات عبر الهدف فئات وبالتالي فإن الدقة ودرجة F1 متساويتان تقريبًا.
عندما تكون وسيطة cv عددًا صحيحًا، يستخدم cross_val_score
استراتيجيات KFold أو StratifiedKFold افتراضيًا، والأخيرة
يتم استخدامه إذا كان المقدر مشتقًا من ClassifierMixin.
من الممكن أيضًا استخدام استراتيجيات التحقق المتبادل الأخرى عن طريق تمرير تقاطع مكرر التحقق من الصحة بدلاً من ذلك، على سبيل المثال:
>>> from sklearn.model_selection import ShuffleSplit
>>> n_samples = X.shape[0]
>>> cv = ShuffleSplit(n_splits=5, test_size=0.3, random_state=0)
>>> cross_val_score(clf, X, y, cv=cv)
array([0.977..., 0.977..., 1. ..., 0.955..., 1. ])
خيار آخر هو استخدام تكرار ينتج عنه انقسامات (تدريب، اختبار) كمصفوفات المؤشرات، على سبيل المثال:
>>> def custom_cv_2folds(X):
... n = X.shape[0]
... i = 1
... while i <= 2:
... idx = np.arange(n * (i - 1) / 2, n * i / 2, dtype=int)
... yield idx, idx
... i += 1
...
>>> custom_cv = custom_cv_2folds(X)
>>> cross_val_score(clf, X, y, cv=custom_cv)
array([1. , 0.973...])
تحويل البيانات مع البيانات المحجوزة#
تمامًا كما هو الحال من المهم اختبار متنبئ على البيانات المحجوزة من التدريب، المعالجة المسبقة (مثل التوحيد القياسي، اختيار الميزات، إلخ.) و تحويلات البيانات المماثلة بالمثل يجب يتم تعلمها من مجموعة التدريب وتطبيقها على البيانات المحجوزة للتنبؤ:
>>> from sklearn import preprocessing
>>> X_train, X_test, y_train, y_test = train_test_split(
... X, y, test_size=0.4, random_state=0)
>>> scaler = preprocessing.StandardScaler().fit(X_train)
>>> X_train_transformed = scaler.transform(X_train)
>>> clf = svm.SVC(C=1).fit(X_train_transformed, y_train)
>>> X_test_transformed = scaler.transform(X_test)
>>> clf.score(X_test_transformed, y_test)
0.9333...
Pipeline يجعل من السهل تكوين
المقدرات، وتوفير هذا السلوك تحت التحقق المتبادل:
>>> from sklearn.pipeline import make_pipeline
>>> clf = make_pipeline(preprocessing.StandardScaler(), svm.SVC(C=1))
>>> cross_val_score(clf, X, y, cv=cv)
array([0.977..., 0.933..., 0.955..., 0.933..., 0.977...])
3.1.1.1. دالة cross_validate وتقييم المقاييس المتعددة#
تختلف دالة cross_validate عن cross_val_score في
طريقتان:
يسمح بتحديد مقاييس متعددة للتقييم.
تُرجع dict تحتوي على أوقات الملاءمة، وأوقات النتيجة (واختياريًا درجات التدريب، والمقدرات المناسبة، ومؤشرات تقسيم القطار والاختبار) بالإضافة إلى درجة الاختبار.
لتقييم مقياس واحد، حيث تكون معلمة التسجيل عبارة عن سلسلة،
قابل للاستدعاء أو لا شيء، ستكون المفاتيح - ['test_score', 'fit_time', 'score_time']
ولتقييم المقاييس المتعددة، تكون قيمة الإرجاع dict مع
المفاتيح التالية -
['test_<scorer1_name>', 'test_<scorer2_name>', 'test_<scorer...>', 'fit_time', 'score_time']
يتم تعيين return_train_score على False افتراضيًا لتوفير وقت الحساب.
لتقييم الدرجات على مجموعة التدريب أيضًا، تحتاج إلى تعيينها على
True. يمكنك أيضًا الاحتفاظ بالمقدر المناسب في كل مجموعة تدريب بواسطة
تعيين return_estimator=True. وبالمثل، يمكنك تعيين
return_indices=True للاحتفاظ بمؤشرات التدريب والاختبار المستخدمة للتقسيم
مجموعة البيانات إلى مجموعات التدريب والاختبار لكل تقسيم cv.
يمكن تحديد المقاييس المتعددة إما كقائمة أو مجموعة أو مجموعة أسماء المسجل المحددة مسبقًا:
>>> from sklearn.model_selection import cross_validate
>>> from sklearn.metrics import recall_score
>>> scoring = ['precision_macro', 'recall_macro']
>>> clf = svm.SVC(kernel='linear', C=1, random_state=0)
>>> scores = cross_validate(clf, X, y, scoring=scoring)
>>> sorted(scores.keys())
['fit_time', 'score_time', 'test_precision_macro', 'test_recall_macro']
>>> scores['test_recall_macro']
array([0.96..., 1. ..., 0.96..., 0.96..., 1. ])
أو كدالة dict تعيين اسم المسجل إلى دالة تسجيل محددة مسبقًا أو مخصصة:
>>> from sklearn.metrics import make_scorer
>>> scoring = {'prec_macro': 'precision_macro',
... 'rec_macro': make_scorer(recall_score, average='macro')}
>>> scores = cross_validate(clf, X, y, scoring=scoring,
... cv=5, return_train_score=True)
>>> sorted(scores.keys())
['fit_time', 'score_time', 'test_prec_macro', 'test_rec_macro',
'train_prec_macro', 'train_rec_macro']
>>> scores['train_rec_macro']
array([0.97..., 0.97..., 0.99..., 0.98..., 0.98...])
فيما يلي مثال على cross_validate باستخدام مقياس واحد:
>>> scores = cross_validate(clf, X, y,
... scoring='precision_macro', cv=5,
... return_estimator=True)
>>> sorted(scores.keys())
['estimator', 'fit_time', 'score_time', 'test_score']
3.1.1.2. الحصول على التنبؤات عن طريق التحقق المتبادل#
تحتوي الدالة cross_val_predict على واجهة مشابهة لـ
cross_val_score، ولكنها تُرجع، لكل عنصر في الإدخال،
التنبؤ الذي تم الحصول عليه لهذا العنصر عندما كان في مجموعة الاختبار. فقط
يمكن استخدام استراتيجيات التحقق المتبادل التي تعين جميع العناصر إلى مجموعة اختبار مرة واحدة بالضبط
(خلاف ذلك، يتم طرح استثناء).
تحذير
ملاحظة حول الاستخدام غير المناسب لـ cross_val_predict
قد تختلف نتيجة cross_val_predict عن تلك
تم الحصول عليها باستخدام cross_val_score حيث يتم تجميع العناصر في
طرق مختلفة. تأخذ الدالة cross_val_score متوسطًا
عبر طيات التحقق المتبادل، بينما cross_val_predict ببساطة
ترجع التسميات (أو الاحتمالات) من عدة نماذج مميزة
غير مميز. وبالتالي، فإن cross_val_predict ليس مقياسًا مناسبًا
خطأ التعميم.
- دالة
cross_val_predictمناسبة لـ: تصور التنبؤات التي تم الحصول عليها من نماذج مختلفة.
مزج النماذج: عند استخدام تنبؤات مقدر خاضع للإشراف واحد لـ تدريب مقدر آخر في طرق المجموعة.
تم تقديم مكررات التحقق المتبادل المتاحة في ما يلي القسم.
أمثلة
sphx_glr_auto_examples_model_selection/plot_roc_crossval.py،
sphx_glr_auto_examples_feature_selection/plot_rfe_with_cross_validation.py،
sphx_glr_auto_examples_model_selection/plot_grid_search_digits.py،
sphx_glr_auto_examples_model_selection/plot_grid_search_text_feature_extraction.py،
sphx_glr_auto_examples_model_selection/plot_cv_predict.py،
sphx_glr_auto_examples_model_selection/plot_nested_cross_validation_iris.py.
3.1.2. مكررات التحقق المتبادل#
تسرد الأقسام التالية الأدوات المساعدة لإنشاء مؤشرات التي يمكن استخدامها لإنشاء تقسيمات مجموعة البيانات وفقًا لاختلاف التقاطع استراتيجيات التحقق من الصحة.
3.1.2.1. مكررات التحقق المتبادل لبيانات i.i.d.#
بافتراض أن بعض البيانات مستقلة وموزعة بشكل متماثل (i.i.d.) هو الافتراض بأن جميع العينات تنبع من نفس عملية التوليد ويفترض أن عملية التوليد ليس لها ذاكرة للعينات التي تم إنشاؤها في الماضي.
يمكن استخدام أدوات التحقق المتبادل التالية في مثل هذه الحالات.
ملاحظة
بينما تعد بيانات i.i.d. افتراضًا شائعًا في نظرية التعلم الآلي، إلا أنها نادرًا ما تحدث يصمد في الممارسة. إذا كان المرء يعرف أن العينات قد تم إنشاؤها باستخدام عملية تعتمد على الوقت، فمن الآمن استخدم مخطط التحقق المتبادل للسلاسل الزمنية. وبالمثل، إذا كنا نعلم أن عملية التوليد لها بنية جماعية (عينات تم جمعها من مواضيع مختلفة، تجارب، قياس الأجهزة)، فمن الآمن استخدام التحقق المتبادل على مستوى المجموعة.
3.1.2.1.1. K-fold#
يقسم KFold جميع العينات في \(k\) مجموعات من العينات،
تسمى الطيات (إذا كان \(k = n\)، فهذا يعادل Leave One
Out استراتيجية)، من أحجام متساوية (إن أمكن). وظيفة التنبؤ هي
تم تعلمه باستخدام \(k - 1\) طيات، ويتم استخدام الطية المتروكة للاختبار.
مثال على التحقق المتبادل من 2-fold على مجموعة بيانات تحتوي على 4 عينات:
>>> import numpy as np
>>> from sklearn.model_selection import KFold
>>> X = ["a", "b", "c", "d"]
>>> kf = KFold(n_splits=2)
>>> for train, test in kf.split(X):
... print("%s %s" % (train, test))
[2 3] [0 1]
[0 1] [2 3]
فيما يلي تصور لسلوك التحقق المتبادل. لاحظ أن
KFold لا يتأثر بالفئات أو المجموعات.
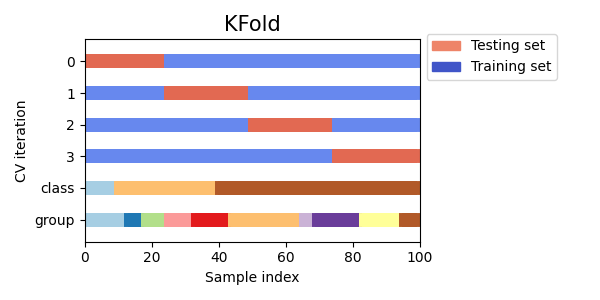
تتكون كل طية من مصفوفتين: الأولى مرتبطة بـ مجموعة التدريب، والثانية إلى مجموعة الاختبار. وبالتالي، يمكن للمرء إنشاء مجموعات التدريب / الاختبار باستخدام فهرسة numpy:
>>> X = np.array([[0., 0.], [1., 1.], [-1., -1.], [2., 2.]])
>>> y = np.array([0, 1, 0, 1])
>>> X_train, X_test, y_train, y_test = X[train], X[test], y[train], y[test]
3.1.2.1.2. Repeated K-Fold#
RepeatedKFold يكرر K-Fold n مرات. يمكن استخدامه عندما يكون المرء
يتطلب تشغيل KFold n مرات، مما ينتج عنه انقسامات مختلفة في
كل تكرار.
مثال على 2-fold K-Fold تم تكراره مرتين:
>>> import numpy as np
>>> from sklearn.model_selection import RepeatedKFold
>>> X = np.array([[1, 2], [3, 4], [1, 2], [3, 4]])
>>> random_state = 12883823
>>> rkf = RepeatedKFold(n_splits=2, n_repeats=2, random_state=random_state)
>>> for train, test in rkf.split(X):
... print("%s %s" % (train, test))
...
[2 3] [0 1]
[0 1] [2 3]
[0 2] [1 3]
[1 3] [0 2]
وبالمثل، يكرر RepeatedStratifiedKFold Stratified K-Fold n مرات
مع عشوائية مختلفة في كل تكرار.
3.1.2.1.3. Leave One Out (LOO)#
LeaveOneOut (أو LOO) هو تحقق متبادل بسيط. كل تعلم
يتم إنشاء المجموعة عن طريق أخذ جميع العينات باستثناء واحدة، ومجموعة الاختبار هي
العينة المتروكة. وبالتالي، لـ \(n\) عينات، لدينا \(n\) مختلف
مجموعات التدريب و \(n\) مجموعة اختبارات مختلفة. هذا الصليب-
لا يهدر إجراء التحقق من الصحة الكثير من البيانات حيث تتم إزالة عينة واحدة فقط من
مجموعة التدريب:
>>> from sklearn.model_selection import LeaveOneOut
>>> X = [1, 2, 3, 4]
>>> loo = LeaveOneOut()
>>> for train, test in loo.split(X):
... print("%s %s" % (train, test))
[1 2 3] [0]
[0 2 3] [1]
[0 1 3] [2]
[0 1 2] [3]
يجب على المستخدمين المحتملين لـ LOO لاختيار النموذج أن يزنوا بعض المحاذير المعروفة. عند مقارنتها بالتحقق المتبادل \(k\)-fold، يبني المرء \(n\) نماذج من \(n\) عينات بدلاً من \(k\) نماذج، حيث \(n > k\). علاوة على ذلك، يتم تدريب كل منها على \(n - 1\) عينات بدلاً من \((k-1) n / k\). في كلتا الحالتين، بافتراض أن \(k\) ليست كبيرة جدًا و \(k < n\)، فإن LOO أكثر تكلفة من الناحية الحسابية من التحقق المتبادل \(k\)-fold.
من حيث الدقة، غالبًا ما ينتج عن LOO تباين كبير كمقدر لـ خطأ الاختبار. بشكل حدسي، منذ \(n - 1\) من يتم استخدام \(n\) عينات لبناء كل نموذج، النماذج التي تم إنشاؤها من الطيات متطابقة تقريبًا مع بعضها البعض ومع النموذج المبني من مجموعة التدريب بأكملها.
ومع ذلك، إذا كان منحنى التعلم حادًا لحجم التدريب المعني، ثم يمكن أن يؤدي التحقق المتبادل من 5 أو 10 طيات إلى المبالغة في تقدير خطأ التعميم.
كقاعدة عامة، يقترح معظم المؤلفين، والأدلة التجريبية، أن 5 أو 10 يجب تفضيل التحقق المتبادل للطي على LOO.
المراجع#
http://www.faqs.org/faqs/ai-faq/neural-nets/part3/section-12.html؛
T. Hastie، R. Tibshirani، J. Friedman، The Elements of Statistical Learning، Springer 2009
L. Breiman، P. Spector Submodel selection and evaluation in regression: The X-random case، International Statistical Review 1992؛
R. Kohavi، A Study of Cross-Validation and Bootstrap for Accuracy Estimation and Model Selection، Intl. Jnt. Conf. AI
R. Bharat Rao، G. Fung، R. Rosales، On the Dangers of Cross-Validation. An Experimental Evaluation، SIAM 2008؛
G. James، D. Witten، T. Hastie، R Tibshirani، An Introduction to Statistical Learning، Springer 2013.
3.1.2.1.4. Leave P Out (LPO)#
LeavePOut يشبه جدًا LeaveOneOut لأنه ينشئ كل
أزواج التدريب / الاختبار الممكنة عن طريق إزالة \(p\) عينات من الكاملة
جلس. لـ \(n\) عينات، ينتج عن هذا \({n \choose p}\) قطار-اختبار
أزواج. على عكس LeaveOneOut و KFold، مجموعات الاختبار ستكون
تتداخل لـ \(p > 1\).
مثال على Leave-2-Out على مجموعة بيانات تحتوي على 4 عينات:
>>> from sklearn.model_selection import LeavePOut
>>> X = np.ones(4)
>>> lpo = LeavePOut(p=2)
>>> for train, test in lpo.split(X):
... print("%s %s" % (train, test))
[2 3] [0 1]
[1 3] [0 2]
[1 2] [0 3]
[0 3] [1 2]
[0 2] [1 3]
[0 1] [2 3]
3.1.2.1.5. تباديل عشوائية للتحقق المتبادل المعروف أيضًا باسم Shuffle & Split#
سيولد مكرر ShuffleSplit عددًا محددًا من قبل المستخدم
انقسامات مجموعة بيانات التدريب / الاختبار المستقلة. يتم خلط العينات أولاً و
ثم تقسم إلى زوج من مجموعات التدريب والاختبار.
من الممكن التحكم في العشوائية لإعادة إنتاج
النتائج عن طريق زرع random_state بشكل صريح عشوائي زائف
مولد الأرقام.
فيما يلي مثال على الاستخدام:
>>> from sklearn.model_selection import ShuffleSplit
>>> X = np.arange(10)
>>> ss = ShuffleSplit(n_splits=5, test_size=0.25, random_state=0)
>>> for train_index, test_index in ss.split(X):
... print("%s %s" % (train_index, test_index))
[9 1 6 7 3 0 5] [2 8 4]
[2 9 8 0 6 7 4] [3 5 1]
[4 5 1 0 6 9 7] [2 3 8]
[2 7 5 8 0 3 4] [6 1 9]
[4 1 0 6 8 9 3] [5 2 7]
فيما يلي تصور لسلوك التحقق المتبادل. لاحظ أن
ShuffleSplit لا يتأثر بالفئات أو المجموعات.
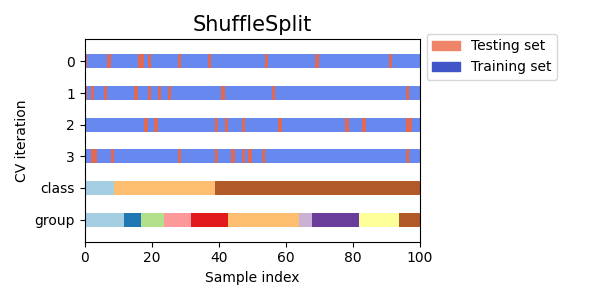
ShuffleSplit هو بالتالي بديل جيد لـ KFold التقاطع
التحقق من الصحة الذي يسمح بالتحكم الدقيق في عدد التكرارات
ونسبة العينات على كل جانب من جوانب تقسيم القطار / الاختبار.
3.1.2.2. مكررات التحقق المتبادل مع التقسيم الطبقي بناءً على تسميات الفئة#
يمكن أن تُظهر بعض مشكلات التصنيف خللًا كبيرًا في التوزيع
من فئات الهدف: على سبيل المثال، يمكن أن يكون هناك عدة مرات سلبية
عينات من العينات الإيجابية. في مثل هذه الحالات، يوصى باستخدام
أخذ العينات الطبقية كما هو مطبق في StratifiedKFold و
StratifiedShuffleSplit لضمان أن ترددات الفئة النسبية هي
يتم الحفاظ عليها تقريبًا في كل طية تدريب والتحقق من الصحة.
3.1.2.2.1. k-fold الطبقي#
StratifiedKFold هو نوع مختلف من k-fold الذي يُرجع طبقات
الطيات: تحتوي كل مجموعة على نفس النسبة المئوية تقريبًا من عينات كل منها
فئة الهدف كمجموعة كاملة.
فيما يلي مثال على التحقق المتبادل الطبقي من 3 طيات على مجموعة بيانات تحتوي على 50 عينة من
فئتين غير متوازنتين. نعرض عدد العينات في كل فئة ونقارنها بـ
KFold.
>>> from sklearn.model_selection import StratifiedKFold, KFold
>>> import numpy as np
>>> X, y = np.ones((50, 1)), np.hstack(([0] * 45, [1] * 5))
>>> skf = StratifiedKFold(n_splits=3)
>>> for train, test in skf.split(X, y):
... print('train - {} | test - {}'.format(
... np.bincount(y[train]), np.bincount(y[test])))
train - [30 3] | test - [15 2]
train - [30 3] | test - [15 2]
train - [30 4] | test - [15 1]
>>> kf = KFold(n_splits=3)
>>> for train, test in kf.split(X, y):
... print('train - {} | test - {}'.format(
... np.bincount(y[train]), np.bincount(y[test])))
train - [28 5] | test - [17]
train - [28 5] | test - [17]
train - [34] | test - [11 5]
يمكننا أن نرى أن StratifiedKFold يحافظ على نسب الفئة
(حوالي 1/10) في كل من مجموعة بيانات التدريب والاختبار.
فيما يلي تصور لسلوك التحقق المتبادل.
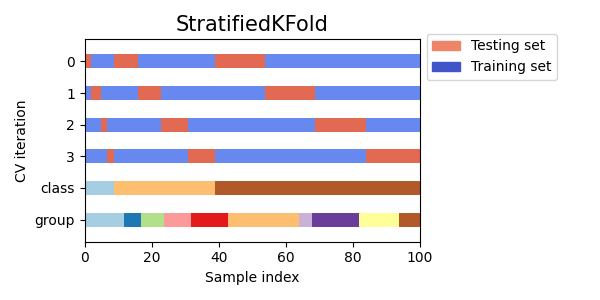
يمكن استخدام RepeatedStratifiedKFold لتكرار Stratified K-Fold n مرات
مع عشوائية مختلفة في كل تكرار.
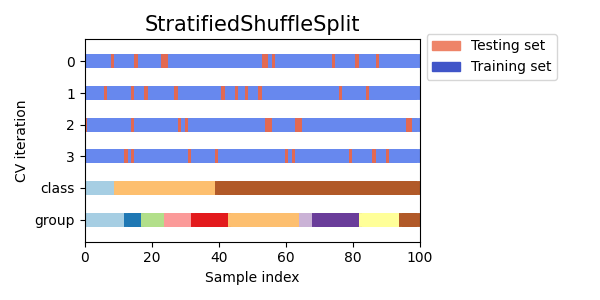
3.1.2.2.2. تقسيم خلط طبقي#
StratifiedShuffleSplit هو نوع مختلف من ShuffleSplit، والذي يُرجع
انقسامات طبقية، على سبيل المثال التي تنشئ انقسامات عن طريق الحفاظ على نفس الشيء
النسبة المئوية لكل فئة هدف كما في المجموعة الكاملة.
فيما يلي تصور لسلوك التحقق المتبادل.
3.1.2.3. تقسيمات الطيات / مجموعات التحقق من الصحة المحددة مسبقًا#
بالنسبة لبعض مجموعات البيانات، يكون التقسيم المحدد مسبقًا للبيانات إلى تدريب-
وطية التحقق من الصحة أو في عدة طيات للتحقق المتبادل بالفعل
موجود. باستخدام PredefinedSplit من الممكن استخدام هذه الطيات
على سبيل المثال عند البحث عن المعلمات الفائقة.
على سبيل المثال، عند استخدام مجموعة التحقق من الصحة، قم بتعيين test_fold على 0 للجميع
العينات التي هي جزء من مجموعة التحقق من الصحة، وإلى -1 لجميع العينات الأخرى.
3.1.2.4. مكررات التحقق المتبادل للبيانات المجمعة#
يتم كسر افتراض i.i.d. إذا كانت عملية التوليد الأساسية تنتج مجموعات من العينات التابعة.
مثل هذا التجميع للبيانات خاص بالمجال. سيكون أحد الأمثلة عندما يكون هناك البيانات الطبية التي تم جمعها من مرضى متعددين، مع أخذ عينات متعددة منكل مريض. ومن المرجح أن تعتمد هذه البيانات على المجموعة الفردية. في مثالنا، سيكون معرف المريض لكل عينة هو معرف مجموعته.
في هذه الحالة، نود أن نعرف ما إذا كان النموذج المدرب على مجموعة معينة من المجموعات تعمم بشكل جيد على المجموعات غير المرئية. لقياس هذا، نحن بحاجة إلى تأكد من أن جميع العينات في طية التحقق من الصحة تأتي من مجموعات غير ممثلة على الإطلاق في طية التدريب المقترنة.
يمكن استخدام أدوات تقسيم التحقق المتبادل التالية للقيام بذلك.
يتم تحديد معرف التجميع للعينات عبر groups
معامل.
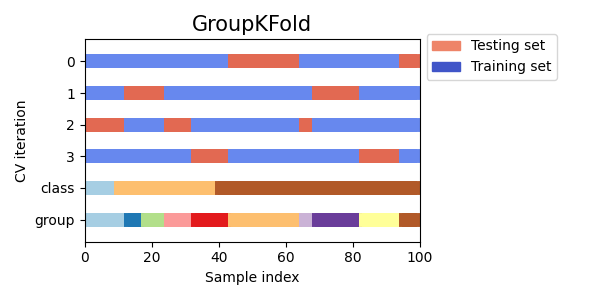
3.1.2.4.1. Group k-fold#
GroupKFold هو نوع مختلف من k-fold يضمن أن نفس المجموعة هي
غير ممثلة في كل من مجموعات الاختبار والتدريب. على سبيل المثال، إذا كانت البيانات
تم الحصول عليها من مواضيع مختلفة مع عدة عينات لكل موضوع وإذا
النموذج مرن بما يكفي للتعلم من ميزات محددة للغاية للشخص
يمكن أن تفشل في التعميم على مواضيع جديدة. GroupKFold يجعل من الممكن
لاكتشاف هذا النوع من حالات الإفراط في التجهيز.
تخيل أن لديك ثلاثة مواضيع، لكل منها رقم مرتبط من 1 إلى 3:
>>> from sklearn.model_selection import GroupKFold
>>> X = [0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 8.8, 9, 10]
>>> y = ["a", "b", "b", "b", "c", "c", "c", "d", "d", "d"]
>>> groups = [1, 1, 1, 2, 2, 2, 3, 3, 3, 3]
>>> gkf = GroupKFold(n_splits=3)
>>> for train, test in gkf.split(X, y, groups=groups):
... print("%s %s" % (train, test))
[0 1 2 3 4 5] [6 7 8 9]
[0 1 2 6 7 8 9] [3 4 5]
[3 4 5 6 7 8 9] [0 1 2]
كل موضوع في طية اختبار مختلفة، ونفس الموضوع ليس أبدًا في
كل من الاختبار والتدريب. لاحظ أن الطيات ليس لها نفس الشيء بالضبط
الحجم بسبب عدم التوازن في البيانات. إذا كان يجب موازنة نسب الفئة
عبر الطيات، StratifiedGroupKFold هو خيار أفضل.
فيما يلي تصور لسلوك التحقق المتبادل.
على غرار KFold، ستشكل مجموعات الاختبار من GroupKFold a
تقسيم كامل لجميع البيانات. على عكس KFold، GroupKFold
غير عشوائي على الإطلاق، بينما يكون KFold عشوائيًا عندما
shuffle=True.
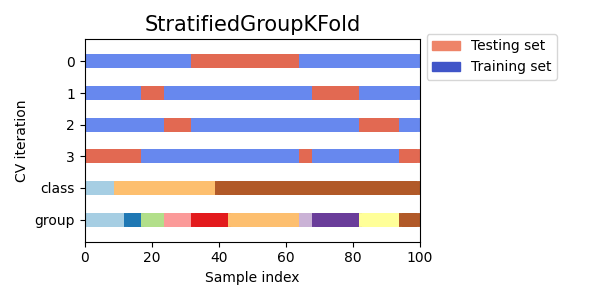
3.1.2.4.2. StratifiedGroupKFold#
StratifiedGroupKFold هو مخطط تحقق متبادل يجمع بين كليهما
StratifiedKFold و GroupKFold. الفكرة هي محاولة
الحفاظ على توزيع الفئات في كل تقسيم مع الحفاظ على كل مجموعة
ضمن تقسيم واحد. قد يكون ذلك مفيدًا عندما يكون لديك غير متوازن
مجموعة بيانات بحيث يؤدي استخدام GroupKFold فقط إلى إنتاج انقسامات منحرفة.
مثال:
>>> from sklearn.model_selection import StratifiedGroupKFold
>>> X = list(range(18))
>>> y = [1] * 6 + [0] * 12
>>> groups = [1, 2, 3, 3, 4, 4, 1, 1, 2, 2, 3, 4, 5, 5, 5, 6, 6, 6]
>>> sgkf = StratifiedGroupKFold(n_splits=3)
>>> for train, test in sgkf.split(X, y, groups=groups):
... print("%s %s" % (train, test))
[ 0 2 3 4 5 6 7 10 11 15 16 17] [ 1 8 9 12 13 14]
[ 0 1 4 5 6 7 8 9 11 12 13 14] [ 2 3 10 15 16 17]
[ 1 2 3 8 9 10 12 13 14 15 16 17] [ 0 4 5 6 7 11]
ملاحظات التنفيذ#
مع التنفيذ الحالي، لا يمكن إجراء خلط كامل في معظم السيناريوهات. عندما shuffle=True، يحدث ما يلي:
يتم خلط جميع المجموعات.
يتم فرز المجموعات حسب الانحراف المعياري للفئات باستخدام الفرز المستقر.
يتم تكرار المجموعات التي تم فرزها وتعيينها إلى طيات.
هذا يعني أنه سيتم خلط المجموعات ذات الانحراف المعياري نفسه لتوزيع الفئة فقط، مما قد يكون مفيدًا عندما تحتوي كل مجموعة على فئة واحدة فقط.
تقوم الخوارزمية بتعيين كل مجموعة بشكل جشع إلى إحدى مجموعات اختبار n_splits، اختيار مجموعة الاختبار التي تقلل من التباين في توزيع الفئة عبر مجموعات الاختبار. يبدأ تعيين المجموعة من المجموعات ذات أعلى إلى أدنى تباين في تكرار الفئة، أي مجموعات كبيرة بلغت ذروتها في واحد أو قليل يتم تعيين الفئات أولاً.
هذا التقسيم دون المستوى الأمثل بمعنى أنه قد ينتج عنه انقسامات غير متوازنة حتى لو كان التقسيم الطبقي المثالي ممكنًا. إذا كان لديك قريب نسبيًا توزيع الفئات في كل مجموعة، فإن استخدام
GroupKFoldأفضل.
فيما يلي تصور لسلوك التحقق المتبادل للمجموعات غير المتساوية:
3.1.2.4.3. Leave One Group Out#
LeaveOneGroupOut هو مخطط للتحقق المتبادل حيث يحتفظ كل تقسيم
العينات التي تنتمي إلى مجموعة معينة. معلومات المجموعة هي
يتم توفيره من خلال مصفوفة تقوم بترميز مجموعة كل عينة.
وبالتالي، تتكون كل مجموعة تدريب من جميع العينات باستثناء تلك
المتعلقة بمجموعة محددة. هذا هو نفسه LeavePGroupsOut مع
n_groups=1 وهو نفس GroupKFold مع n_splits يساوي
عدد التسميات الفريدة التي تم تمريرها إلى معلمة groups.
على سبيل المثال، في حالات التجارب المتعددة، LeaveOneGroupOut
يمكن استخدامها لإنشاء تحقق متبادل بناءً على التجارب المختلفة:
نقوم بإنشاء مجموعة تدريب باستخدام عينات جميع التجارب باستثناء واحدة:
>>> from sklearn.model_selection import LeaveOneGroupOut
>>> X = [1, 5, 10, 50, 60, 70, 80]
>>> y = [0, 1, 1, 2, 2, 2, 2]
>>> groups = [1, 1, 2, 2, 3, 3, 3]
>>> logo = LeaveOneGroupOut()
>>> for train, test in logo.split(X, y, groups=groups):
... print("%s %s" % (train, test))
[2 3 4 5 6] [0 1]
[0 1 4 5 6] [2 3]
[0 1 2 3] [4 5 6]
تطبيق شائع آخر هو استخدام معلومات الوقت: على سبيل المثال يمكن أن تكون المجموعات هي سنة جمع العينات وبالتالي تسمح للتحقق المتبادل مقابل الانقسامات القائمة على الوقت.
3.1.2.4.4. Leave P Groups Out#
LeavePGroupsOut مشابه لـ LeaveOneGroupOut، لكنه يزيل
عينات متعلقة بـ \(P\) مجموعات لكل مجموعة تدريب / اختبار. كل ما هو ممكن
يتم استبعاد مجموعات \(P\)، مما يعني أن مجموعات الاختبار ستتداخل
لـ \(P>1\).
مثال على Leave-2-Group Out:
>>> from sklearn.model_selection import LeavePGroupsOut
>>> X = np.arange(6)
>>> y = [1, 1, 1, 2, 2, 2]
>>> groups = [1, 1, 2, 2, 3, 3]
>>> lpgo = LeavePGroupsOut(n_groups=2)
>>> for train, test in lpgo.split(X, y, groups=groups):
... print("%s %s" % (train, test))
[4 5] [0 1 2 3]
[2 3] [0 1 4 5]
[0 1] [2 3 4 5]
3.1.2.4.5. Group Shuffle Split#
يتصرف مكرر GroupShuffleSplit كمزيج من
ShuffleSplit و LeavePGroupsOut، ويولد
سلسلة من الأقسام العشوائية التي تكون فيها مجموعة فرعية من المجموعات
يتم الاحتفاظ بها لكل تقسيم. يتم تنفيذ كل تقسيم تدريب / اختبار بشكل مستقل بمعنى
لا توجد علاقة مضمونة بين مجموعات الاختبار المتتالية.
فيما يلي مثال على الاستخدام:
>>> from sklearn.model_selection import GroupShuffleSplit
>>> X = [0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 0.001]
>>> y = ["a", "b", "b", "b", "c", "c", "c", "a"]
>>> groups = [1, 1, 2, 2, 3, 3, 4, 4]
>>> gss = GroupShuffleSplit(n_splits=4, test_size=0.5, random_state=0)
>>> for train, test in gss.split(X, y, groups=groups):
... print("%s %s" % (train, test))
...
[0 1 2 3] [4 5 6 7]
[2 3 6 7] [0 1 4 5]
[2 3 4 5] [0 1 6 7]
[4 5 6 7] [0 1 2 3]
فيما يلي تصور لسلوك التحقق المتبادل.
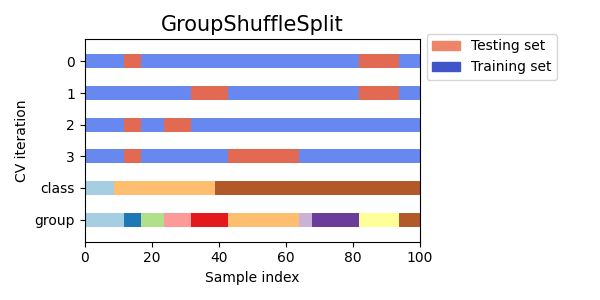
هذه الفئة مفيدة عندما يكون سلوك LeavePGroupsOut هو
المرغوب فيه، لكن عدد المجموعات كبير بما يكفي لتوليد كل
ستكون الأقسام الممكنة مع حجب مجموعات \(P\) باهظة الثمن.
في مثل هذا السيناريو، يوفر GroupShuffleSplit
عينة عشوائية (مع الاستبدال) من تقسيمات القطار / الاختبار
تم إنشاؤها بواسطة LeavePGroupsOut.
3.1.2.5. استخدام مكررات التحقق المتبادل لتقسيم التدريب والاختبار#
قد تكون وظائف التحقق المتبادل للمجموعة المذكورة أعلاه مفيدة أيضًا لتقسيم
مجموعة بيانات إلى مجموعات فرعية للتدريب والاختبار. لاحظ أن الراحة
الدالة train_test_split هي غلاف حول ShuffleSplit
وبالتالي يسمح فقط بالتقسيم الطبقي (باستخدام تسميات الفئة)
ولا يمكن أن تمثل المجموعات.
لإجراء تقسيم التدريب والاختبار، استخدم المؤشرات للتدريب والاختبار
المجموعات الفرعية التي ينتجها المولد بواسطة طريقة split() لـ
مقسم التحقق المتبادل. فمثلا:
>>> import numpy as np
>>> from sklearn.model_selection import GroupShuffleSplit
>>> X = np.array([0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 0.001])
>>> y = np.array(["a", "b", "b", "b", "c", "c", "c", "a"])
>>> groups = np.array([1, 1, 2, 2, 3, 3, 4, 4])
>>> train_indx, test_indx = next(
... GroupShuffleSplit(random_state=7).split(X, y, groups)
... )
>>> X_train, X_test, y_train, y_test = \
... X[train_indx], X[test_indx], y[train_indx], y[test_indx]
>>> X_train.shape, X_test.shape
((6,), (2,))
>>> np.unique(groups[train_indx]), np.unique(groups[test_indx])
(array([1, 2, 4]), array([3]))
3.1.2.6. التحقق المتبادل من بيانات السلاسل الزمنية#
تتميز بيانات السلاسل الزمنية بالارتباط بين الملاحظات
التي هي قريبة في الوقت المناسب (الارتباط التلقائي). ومع ذلك، الكلاسيكية
تقنيات التحقق المتبادل مثل KFold و
ShuffleSplit تفترض أن العينات مستقلة و
موزعة بشكل متماثل، وسيؤدي إلى ارتباط غير معقول
بين حالات التدريب والاختبار (ينتج عنها تقديرات ضعيفة لـ
خطأ التعميم) على بيانات السلاسل الزمنية. لذلك، من المهم جدًا
لتقييم نموذجنا لبيانات السلاسل الزمنية على الملاحظات "المستقبلية"
على الأقل مثل تلك التي تُستخدم لتدريب النموذج. لتحقيق هذا، واحد
يتم توفير الحل بواسطة TimeSeriesSplit.
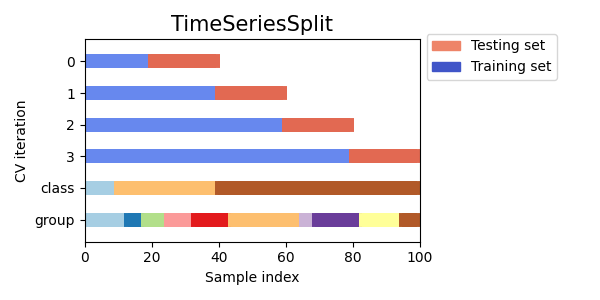
3.1.2.6.1. تقسيم السلاسل الزمنية#
TimeSeriesSplit هو نوع مختلف من k-fold الذي
يعيد أول \(k\) طيات كمجموعة تدريب و \((k+1)\)
طية كمجموعة اختبار. لاحظ أنه على عكس طرق التحقق المتبادل القياسية،
مجموعات التدريب المتتالية هي مجموعات شاملة لتلك التي تأتي قبلها.
أيضًا، يضيف جميع البيانات الفائضة إلى قسم التدريب الأول، والذي
يتم استخدامه دائمًا لتدريب النموذج.
يمكن استخدام هذه الفئة للتحقق المتبادل من عينات بيانات السلاسل الزمنية التي لوحظت على فترات زمنية ثابتة.
مثال على التحقق المتبادل للسلاسل الزمنية المكونة من 3 انقسامات على مجموعة بيانات تحتوي على 6 عينات:
>>> from sklearn.model_selection import TimeSeriesSplit
>>> X = np.array([[1, 2], [3, 4], [1, 2], [3, 4], [1, 2], [3, 4]])
>>> y = np.array([1, 2, 3, 4, 5, 6])
>>> tscv = TimeSeriesSplit(n_splits=3)
>>> print(tscv)
TimeSeriesSplit(gap=0, max_train_size=None, n_splits=3, test_size=None)
>>> for train, test in tscv.split(X):
... print("%s %s" % (train, test))
[0 1 2] [3]
[0 1 2 3] [4]
[0 1 2 3 4] [5]
فيما يلي تصور لسلوك التحقق المتبادل.
3.1.3. ملاحظة حول الخلط#
إذا لم يكن ترتيب البيانات عشوائيًا (على سبيل المثال، عينات بنفس تسمية الفئة متجاورة)، قد يكون خلطها أولاً ضروريًا للحصول على تقاطع ذي مغزى- نتيجة التحقق من الصحة. ومع ذلك، قد يكون العكس صحيحًا إذا كانت العينات ليست كذلك موزعة بشكل مستقل ومتماثل. على سبيل المثال، إذا كانت العينات تتوافق لمقالات إخبارية، ومرتبة حسب وقت نشرها، ثم خلط من المرجح أن تؤدي البيانات إلى نموذج مفرط التجهيز ودرجة تحقق من الصحة متضخمة: سيتم اختباره على عينات متشابهة بشكل مصطنع (قريبة من الوقت) لعينات التدريب.
تحتوي بعض مكررات التحقق المتبادل، مثل KFold، على خيار مدمج
لخلط مؤشرات البيانات قبل تقسيمها. لاحظ أن:
يستهلك هذا ذاكرة أقل من خلط البيانات مباشرة.
افتراضيًا، لا يحدث خلط، بما في ذلك (الطبقي) K fold cross- التحقق من الصحة الذي يتم إجراؤه عن طريق تحديد
cv=some_integerإلىcross_val_score، بحث الشبكة، إلخ. ضع في اعتبارك ذلكtrain_test_splitلا يزال يُرجع تقسيمًا عشوائيًا.معلمة
random_stateافتراضية إلىNone، مما يعني أن سيكون الخلط مختلفًا في كل مرةKFold(..., shuffle=True)هو متكرر. ومع ذلك، سيستخدمGridSearchCVنفس الخلط لكل مجموعة من المعلمات التي تم التحقق من صحتها من خلال استدعاء واحد لطريقةfit.للحصول على نتائج متطابقة لكل تقسيم، قم بتعيين
random_stateإلى عدد صحيح.
لمزيد من التفاصيل حول كيفية التحكم في عشوائية مقسمات السيرة الذاتية وتجنب المزالق الشائعة، انظر التحكم في العشوائية.
3.1.4. التحقق المتبادل واختيار النموذج#
يمكن أيضًا استخدام مكررات التحقق المتبادل لأداء النموذج مباشرةً الاختيار باستخدام Grid Search للمعلمات الفائقة المثلى لـ النموذج. هذا هو موضوع القسم التالي: ضبط المعلمات الفائقة لمُقدِّر.
3.1.5. درجة اختبار التقليب#
يقدم permutation_test_score طريقة أخرى
لتقييم أداء المصنفات. يوفر التقليب القائم على
القيمة الاحتمالية، والتي تمثل مدى احتمالية أداء ملحوظ لـ
سيتم الحصول على المصنف عن طريق الصدفة. الفرضية الصفرية في هذا الاختبار هي
أن المصنف يفشل في الاستفادة من أي اعتماد إحصائي بين
الميزات والتسميات لإجراء تنبؤات صحيحة على البيانات المتروكة.
permutation_test_score يولد فارغًا
التوزيع عن طريق حساب n_permutations تباديل مختلفة لـ
البيانات. في كل تبديل، يتم خلط التسميات بشكل عشوائي، وبالتالي إزالة
أي اعتماد بين الميزات والتسميات. إخراج القيمة الاحتمالية
هو جزء التباديل الذي يكون متوسط درجة التحقق المتبادل
التي تم الحصول عليها بواسطة النموذج أفضل من درجة التحقق المتبادل التي تم الحصول عليها بواسطة
النموذج باستخدام البيانات الأصلية. للحصول على نتائج موثوقة n_permutations
يجب أن تكون عادةً أكبر من 100 و cv بين 3-10 طيات.
توفر القيمة الاحتمالية المنخفضة دليلًا على أن مجموعة البيانات تحتوي على اعتماد حقيقي بين الميزات والتسميات وكان المصنف قادرًا على استخدام هذا للحصول على نتائج جيدة. يمكن أن تكون القيمة الاحتمالية العالية بسبب نقص الاعتماد بين الميزات والتسميات (لا يوجد فرق في قيم الميزات بين الفئات) أو لأن المصنف لم يكن قادرًا على استخدام التبعية في البيانات. في الحالة الأخيرة، باستخدام مصنف أكثر ملاءمة قادر على استخدام الهيكل في البيانات، سينتج عنه أقل القيمة الاحتمالية.
يوفر التحقق المتبادل معلومات حول مدى جودة تعميم المصنف،
على وجه التحديد نطاق الأخطاء المتوقعة للمصنف. ومع ذلك،
لا يزال من الممكن أن يؤدي المصنف المدرب على مجموعة بيانات عالية الأبعاد بدون بنية
أداء أفضل من المتوقع في التحقق المتبادل، عن طريق الصدفة.
يمكن أن يحدث هذا عادةً مع مجموعات البيانات الصغيرة التي تحتوي على أقل من بضع مئات
عينات.
permutation_test_score يوفر معلومات
حول ما إذا كان المصنف قد وجد بنية فئة حقيقية ويمكن أن يساعد في
تقييم أداء المصنف.
من المهم ملاحظة أن هذا الاختبار قد ثبت أنه ينتج منخفضًا القيم الاحتمالية حتى لو كان هناك هيكل ضعيف فقط في البيانات لأن في مجموعات البيانات المتبادلة المقابلة لا يوجد هيكل على الإطلاق. هذا لذلك، فإن الاختبار قادر فقط على إظهار متى يتفوق النموذج بشكل موثوق التخمين العشوائي.
أخيرًا، يتم حساب permutation_test_score
باستخدام القوة الغاشمة وملاءمة (n_permutations + 1) * n_cv نماذج داخليًا.
لذلك لا يمكن تتبعه إلا مع مجموعات البيانات الصغيرة التي تناسب
نموذج فردي سريع جدًا.
أمثلة
sphx_glr_auto_examples_model_selection/plot_permutation_tests_for_classification.py
المراجع#
Ojala and Garriga. Permutation Tests for Studying Classifier Performance. J. Mach. Learn. Res. 2010.
