ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
نمذجة توزيع الأنواع#
نمذجة التوزيعات الجغرافية للأنواع هي مشكلة مهمة
في علم الأحياء الحفاظي. في هذا المثال، نقوم
بنمذجة التوزيع الجغرافي لثدييين من أمريكا الجنوبية
بناءً على الملاحظات السابقة و14 متغيرًا بيئيًا. نظرًا لأن لدينا فقط أمثلة إيجابية (لا توجد ملاحظات غير ناجحة), فإننا نطرح هذه المشكلة كتقدير كثافة ونستخدم OneClassSVM
كأداة النمذجة الخاصة بنا. توفر مجموعة البيانات من قبل فيليبس وآخرون. (2006).
إذا كان متاحًا، يستخدم المثال
basemap
لرسم خطوط السواحل والحدود الوطنية لأمريكا الجنوبية.
النوعان هما:
Bradypus variegatus، الكسلان ذو الحلق البني.
Microryzomys minutus، المعروف أيضًا باسم فأر الأرز الصغير الغابوي، وهو قارض يعيش في بيرو، كولومبيا، الإكوادور، بيرو، وفنزويلا.
مراجع#
"نمذجة الحد الأقصى من الإنتروبيا لتوزيعات الأنواع الجغرافية" س. ج. فيليبس، ر. ب. أندرسون، ر. إي. شابير - النمذجة البيئية، 190:231-259، 2006.
_ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _
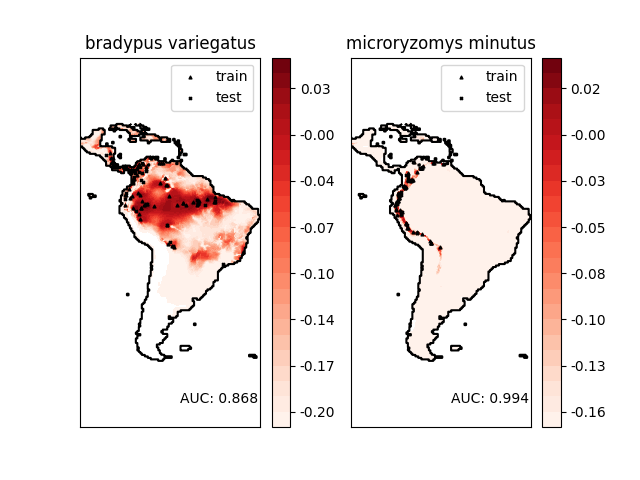
نمذجة توزيع النوع 'bradypus variegatus'
- fit OneClassSVM ... تم.
- plot coastlines from coverage
- predict species distribution
Area under the ROC curve : 0.868443
_ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _ _
نمذجة توزيع النوع 'microryzomys minutus'
- fit OneClassSVM ... تم.
- plot coastlines from coverage
- predict species distribution
Area under the ROC curve : 0.993919
time elapsed: 6.10s
# المؤلفون: مطوري سكايلرن
# معرف الترخيص: BSD-3-Clause
from time import time
import matplotlib.pyplot as plt
import numpy as np
from sklearn import metrics, svm
from sklearn.datasets import fetch_species_distributions
from sklearn.utils import Bunch
# إذا كان basemap متاحًا، فسنستخدمه.
# وإلا، فسوف نرتجل لاحقًا...
try:
from mpl_toolkits.basemap import Basemap
basemap = True
except ImportError:
basemap = False
def construct_grids(batch):
"""بناء شبكة الخريطة من كائن الدفعة
المعلمات
----------
batch : Batch object
الكائن الذي تم إرجاعه بواسطة :func:`fetch_species_distributions`
الإرجاع
-------
(xgrid, ygrid) : 1-D arrays
الشبكة المقابلة للقيم في batch.coverages
"""
# إحداثيات x,y لخلايا الزاوية
xmin = batch.x_left_lower_corner + batch.grid_size
xmax = xmin + (batch.Nx * batch.grid_size)
ymin = batch.y_left_lower_corner + batch.grid_size
ymax = ymin + (batch.Ny * batch.grid_size)
# إحداثيات x لخلايا الشبكة
xgrid = np.arange(xmin, xmax, batch.grid_size)
# إحداثيات y لخلايا الشبكة
ygrid = np.arange(ymin, ymax, batch.grid_size)
return (xgrid, ygrid)
def create_species_bunch(species_name, train, test, coverages, xgrid, ygrid):
"""إنشاء حزمة معلومات حول كائن حي معين
سيستخدم هذا الدالة مصفوفات الاختبار/التدريب لاستخراج
البيانات الخاصة باسم النوع المحدد.
"""
bunch = Bunch(name=" ".join(species_name.split("_")[:2]))
species_name = species_name.encode("ascii")
points = dict(test=test, train=train)
for label, pts in points.items():
# اختيار النقاط المرتبطة بالنوع المطلوب
pts = pts[pts["species"] == species_name]
bunch["pts_%s" % label] = pts
# تحديد قيم التغطية لكل من نقاط التدريب والاختبار
ix = np.searchsorted(xgrid, pts["dd long"])
iy = np.searchsorted(ygrid, pts["dd lat"])
bunch["cov_%s" % label] = coverages[:, -iy, ix].T
return bunch
def plot_species_distribution(
species=("bradypus_variegatus_0", "microryzomys_minutus_0")
):
"""
رسم توزيع الأنواع.
"""
if len(species) > 2:
print(
"ملاحظة: عند توفير أكثر من نوعين،"
" سيتم استخدام النوعين الأولين فقط"
)
t0 = time()
# تحميل البيانات المضغوطة
data = fetch_species_distributions()
# إعداد شبكة البيانات
xgrid, ygrid = construct_grids(data)
# الشبكة في إحداثيات x,y
X, Y = np.meshgrid(xgrid, ygrid[::-1])
# إنشاء حزمة لكل نوع
BV_bunch = create_species_bunch(
species[0], data.train, data.test, data.coverages, xgrid, ygrid
)
MM_bunch = create_species_bunch(
species[1], data.train, data.test, data.coverages, xgrid, ygrid
)
# نقاط الخلفية (إحداثيات الشبكة) للتقييم
np.random.seed(13)
background_points = np.c_[
np.random.randint(low=0, high=data.Ny, size=10000),
np.random.randint(low=0, high=data.Nx, size=10000),
].T
# سنستفيد من حقيقة أن coverages[6] لديها قياسات في جميع
# نقاط اليابسة. سيساعدنا هذا في التمييز بين اليابسة والماء.
land_reference = data.coverages[6]
# تناسب، توقع، ورسم لكل نوع.
for i, species in enumerate([BV_bunch, MM_bunch]):
print("_ " * 80)
print("نمذجة توزيع النوع '%s'" % species.name)
# توحيد الميزات
mean = species.cov_train.mean(axis=0)
std = species.cov_train.std(axis=0)
train_cover_std = (species.cov_train - mean) / std
# توحيد الميزات
mean = species.cov_train.mean(axis=0)
std = species.cov_train.std(axis=0)
train_cover_std = (species.cov_train - mean) / std
# تناسب OneClassSVM
print(" - fit OneClassSVM ... ", end="")
clf = svm.OneClassSVM(nu=0.1, kernel="rbf", gamma=0.5)
clf.fit(train_cover_std)
print("تم.")
# رسم خريطة أمريكا الجنوبية
plt.subplot(1, 2, i + 1)
if basemap:
print(" - plot coastlines using basemap")
m = Basemap(
projection="cyl",
llcrnrlat=Y.min(),
urcrnrlat=Y.max(),
llcrnrlon=X.min(),
urcrnrlon=X.max(),
resolution="c",
)
m.drawcoastlines()
m.drawcountries()
else:
print(" - plot coastlines from coverage")
plt.contour(
X, Y, land_reference, levels=[-9998], colors="k", linestyles="solid"
)
plt.xticks([])
plt.yticks([])
print(" - predict species distribution")
# توقع توزيع الأنواع باستخدام بيانات التدريب
Z = np.ones((data.Ny, data.Nx), dtype=np.float64)
# سنقوم بالتنبؤ فقط لنقاط اليابسة.
idx = np.where(land_reference > -9999)
coverages_land = data.coverages[:, idx[0], idx[1]].T
pred = clf.decision_function((coverages_land - mean) / std)
Z *= pred.min()
Z[idx[0], idx[1]] = pred
levels = np.linspace(Z.min(), Z.max(), 25)
Z[land_reference == -9999] = -9999
# رسم خطوط كفاف التوقع
plt.contourf(X, Y, Z, levels=levels, cmap=plt.cm.Reds)
plt.colorbar(format="%.2f")
# رسم نقاط التدريب/الاختبار
plt.scatter(
species.pts_train["dd long"],
species.pts_train["dd lat"],
s=2**2,
c="black",
marker="^",
label="train",
)
plt.scatter(
species.pts_test["dd long"],
species.pts_test["dd lat"],
s=2**2,
c="black",
marker="x",
label="test",
)
plt.legend()
plt.title(species.name)
plt.axis("equal")
# حساب AUC فيما يتعلق بنقاط الخلفية
pred_background = Z[background_points[0], background_points[1]]
pred_test = clf.decision_function((species.cov_test - mean) / std)
scores = np.r_[pred_test, pred_background]
y = np.r_[np.ones(pred_test.shape), np.zeros(pred_background.shape)]
fpr, tpr, thresholds = metrics.roc_curve(y, scores)
roc_auc = metrics.auc(fpr, tpr)
plt.text(-35, -70, "AUC: %.3f" % roc_auc, ha="right")
print("\n Area under the ROC curve : %f" % roc_auc)
print("\ntime elapsed: %.2fs" % (time() - t0))
plot_species_distribution()
plt.show()
Total running time of the script: (0 minutes 6.258 seconds)
Related examples

رسم أسطح القرار لمجموعات الأشجار على مجموعة بيانات إيريس