ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
تحويل ميزة التجزئة باستخدام الأشجار العشوائية تمامًا#
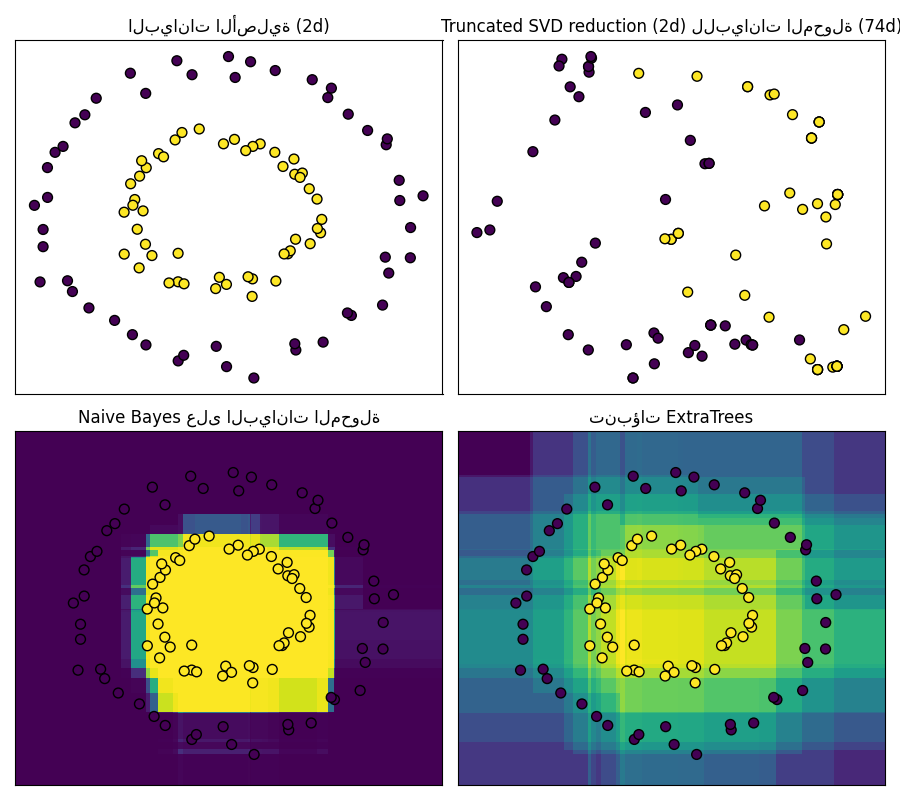
يوفر RandomTreesEmbedding طريقة لتعيين البيانات إلى تمثيل متناثر عالي الأبعاد، والذي قد يكون مفيدًا للتصنيف. التعيين غير خاضع للإشراف تمامًا وفعال للغاية.
يوضح هذا المثال الأقسام التي قدمتها العديد من الأشجار ويوضح كيف يمكن أيضًا استخدام التحويل لتقليل الأبعاد غير الخطي أو التصنيف غير الخطي.
غالبًا ما تشترك النقاط المتجاورة في نفس ورقة الشجرة، وبالتالي تشترك في أجزاء كبيرة من تمثيلها المجزأ. يسمح هذا بفصل دائرتين متحدة المركز ببساطة بناءً على المكونات الأساسية للبيانات المحولة باستخدام SVD المقطوع.
في المساحات عالية الأبعاد، غالبًا ما تحقق المصنفات الخطية دقة ممتازة. بالنسبة للبيانات الثنائية المتناثرة، فإن BernoulliNB مناسب بشكل خاص. تقارن الصف السفلي حد القرار الذي تم الحصول عليه بواسطة BernoulliNB في المساحة المحولة مع غابات ExtraTreesClassifier التي تم تعلمها على البيانات الأصلية.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import make_circles
from sklearn.decomposition import TruncatedSVD
from sklearn.ensemble import ExtraTreesClassifier, RandomTreesEmbedding
from sklearn.naive_bayes import BernoulliNB
# إنشاء مجموعة بيانات اصطناعية
X, y = make_circles(factor=0.5, random_state=0, noise=0.05)
# استخدام RandomTreesEmbedding لتحويل البيانات
hasher = RandomTreesEmbedding(n_estimators=10, random_state=0, max_depth=3)
X_transformed = hasher.fit_transform(X)
# تصور النتيجة بعد تقليل الأبعاد باستخدام SVD المقطوع
svd = TruncatedSVD(n_components=2)
X_reduced = svd.fit_transform(X_transformed)
# تعلم مصنف Naive Bayes على البيانات المحولة
nb = BernoulliNB()
nb.fit(X_transformed, y)
# تعلم ExtraTreesClassifier للمقارنة
trees = ExtraTreesClassifier(max_depth=3, n_estimators=10, random_state=0)
trees.fit(X, y)
# مخطط مبعثر للبيانات الأصلية والمختزلة
fig = plt.figure(figsize=(9, 8))
ax = plt.subplot(221)
ax.scatter(X[:, 0], X[:, 1], c=y, s=50, edgecolor="k")
ax.set_title("البيانات الأصلية (2d)")
ax.set_xticks(())
ax.set_yticks(())
ax = plt.subplot(222)
ax.scatter(X_reduced[:, 0], X_reduced[:, 1], c=y, s=50, edgecolor="k")
ax.set_title(
"Truncated SVD reduction (2d) للبيانات المحولة (%dd)" % X_transformed.shape[1]
)
ax.set_xticks(())
ax.set_yticks(())
# ارسم القرار في المساحة الأصلية. لذلك، سنقوم بتعيين لون
# لكل نقطة في الشبكة [x_min, x_max]x[y_min, y_max].
h = 0.01
x_min, x_max = X[:, 0].min() - 0.5, X[:, 0].max() + 0.5
y_min, y_max = X[:, 1].min() - 0.5, X[:, 1].max() + 0.5
xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h))
# تحويل الشبكة باستخدام RandomTreesEmbedding
transformed_grid = hasher.transform(np.c_[xx.ravel(), yy.ravel()])
y_grid_pred = nb.predict_proba(transformed_grid)[:, 1]
ax = plt.subplot(223)
ax.set_title("Naive Bayes على البيانات المحولة")
ax.pcolormesh(xx, yy, y_grid_pred.reshape(xx.shape))
ax.scatter(X[:, 0], X[:, 1], c=y, s=50, edgecolor="k")
ax.set_ylim(-1.4, 1.4)
ax.set_xlim(-1.4, 1.4)
ax.set_xticks(())
ax.set_yticks(())
# تحويل الشبكة باستخدام ExtraTreesClassifier
y_grid_pred = trees.predict_proba(np.c_[xx.ravel(), yy.ravel()])[:, 1]
ax = plt.subplot(224)
ax.set_title("تنبؤات ExtraTrees")
ax.pcolormesh(xx, yy, y_grid_pred.reshape(xx.shape))
ax.scatter(X[:, 0], X[:, 1], c=y, s=50, edgecolor="k")
ax.set_ylim(-1.4, 1.4)
ax.set_xlim(-1.4, 1.4)
ax.set_xticks(())
ax.set_yticks(())
plt.tight_layout()
plt.show()
Total running time of the script: (0 minutes 0.455 seconds)
Related examples
تغيير معامل التنظيم في الشبكة العصبية متعددة الطبقات
تصنيف العملية الغاوسية (GPC) على مجموعة بيانات iris