ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
أبرز ميزات الإصدار 1.4 من scikit-learn#
يسعدنا الإعلان عن إصدار scikit-learn 1.4! تم إجراء العديد من الإصلاحات والتحسينات، بالإضافة إلى بعض الميزات الرئيسية الجديدة. نستعرض أدناه بعض الميزات الرئيسية لهذا الإصدار. للاطلاع على قائمة شاملة بجميع التغييرات، يرجى الرجوع إلى ملاحظات الإصدار.
لتثبيت أحدث إصدار (باستخدام pip):
pip install --upgrade scikit-learn
أو باستخدام conda:
conda install -c conda-forge scikit-learn
دعم الأنواع الفئوية في HistGradientBoosting بشكل أصلي في DataFrames#
ensemble.HistGradientBoostingClassifier و
ensemble.HistGradientBoostingRegressor يدعمان الآن الأنواع الفئوية بشكل أصلي في أطر البيانات. هنا لدينا مجموعة بيانات تحتوي على مزيج من
الميزات الفئوية والرقمية:
from sklearn.datasets import fetch_openml
X_adult, y_adult = fetch_openml("adult", version=2, return_X_y=True)
# إزالة الأعمدة المكررة وغير المطلوبة
X_adult = X_adult.drop(["education-num", "fnlwgt"], axis="columns")
X_adult.dtypes
age int64
workclass category
education category
marital-status category
occupation category
relationship category
race category
sex category
capital-gain int64
capital-loss int64
hours-per-week int64
native-country category
dtype: object
من خلال تعيين categorical_features="from_dtype"، يقوم مصنف التدرج التدريجي
بمعاملة الأعمدة ذات الأنواع الفئوية على أنها ميزات فئوية في
الخوارزمية:
from sklearn.ensemble import HistGradientBoostingClassifier
from sklearn.model_selection import train_test_split
from sklearn.metrics import roc_auc_score
X_train, X_test, y_train, y_test = train_test_split(X_adult, y_adult, random_state=0)
hist = HistGradientBoostingClassifier(categorical_features="from_dtype")
hist.fit(X_train, y_train)
y_decision = hist.decision_function(X_test)
print(f"ROC AUC score is {roc_auc_score(y_test, y_decision)}")
ROC AUC score is 0.9285339263136636
دعم إخراج Polars في set_output#
تدعم محولات scikit-learn الآن إخراج Polars باستخدام واجهة برمجة التطبيقات set_output.
import polars as pl
from sklearn.preprocessing import StandardScaler
from sklearn.preprocessing import OneHotEncoder
from sklearn.compose import ColumnTransformer
df = pl.DataFrame(
{"height": [120, 140, 150, 110, 100], "pet": ["dog", "cat", "dog", "cat", "cat"]}
)
preprocessor = ColumnTransformer(
[
("numerical", StandardScaler(), ["height"]),
("categorical", OneHotEncoder(sparse_output=False), ["pet"]),
],
verbose_feature_names_out=False,
)
preprocessor.set_output(transform="polars")
df_out = preprocessor.fit_transform(df)
df_out
print(f"Output type: {type(df_out)}")
Output type: <class 'polars.dataframe.frame.DataFrame'>
دعم القيم المفقودة في Random Forest#
تدعم الفئات ensemble.RandomForestClassifier و
ensemble.RandomForestRegressor القيم المفقودة الآن. عند تدريب
كل شجرة فردية، يقوم المقسم بتقييم كل عتبة محتملة مع
القيم المفقودة التي تذهب إلى العقد اليسرى واليمنى. لمزيد من التفاصيل في
دليل المستخدم.
import numpy as np
from sklearn.ensemble import RandomForestClassifier
X = np.array([0, 1, 6, np.nan]).reshape(-1, 1)
y = [0, 0, 1, 1]
forest = RandomForestClassifier(random_state=0).fit(X, y)
forest.predict(X)
array([0, 0, 1, 1])
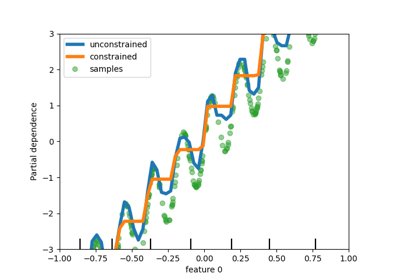
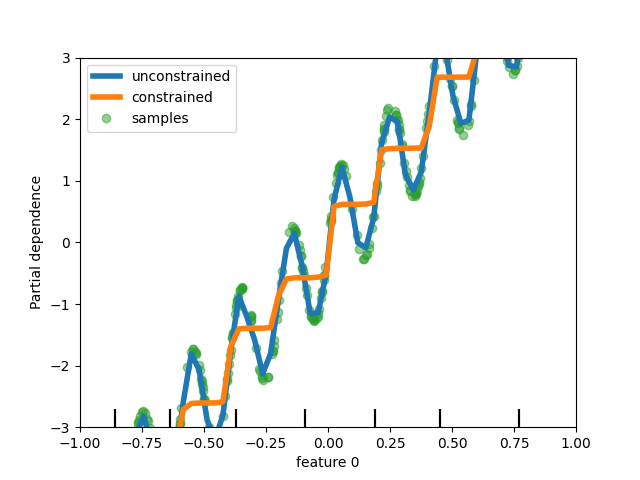
إضافة دعم القيود الأحادية الاتجاه في النماذج القائمة على الشجرة#
على الرغم من أننا أضفنا دعم القيود الأحادية الاتجاه في التدرج التدريجي القائم على الرسم البياني في scikit-learn 0.23، إلا أننا ندعم هذه الميزة الآن لجميع النماذج الأخرى القائمة على الشجرة مثل الأشجار، والغابات العشوائية، والأشجار الإضافية، والتدرج التدريجي الدقيق. هنا، نعرض هذه الميزة للغابة العشوائية في مشكلة الانحدار.
import matplotlib.pyplot as plt
from sklearn.inspection import PartialDependenceDisplay
from sklearn.ensemble import RandomForestRegressor
n_samples = 500
rng = np.random.RandomState(0)
X = rng.randn(n_samples, 2)
noise = rng.normal(loc=0.0, scale=0.01, size=n_samples)
y = 5 * X[:, 0] + np.sin(10 * np.pi * X[:, 0]) - noise
rf_no_cst = RandomForestRegressor().fit(X, y)
rf_cst = RandomForestRegressor(monotonic_cst=[1, 0]).fit(X, y)
disp = PartialDependenceDisplay.from_estimator(
rf_no_cst,
X,
features=[0],
feature_names=["feature 0"],
line_kw={"linewidth": 4, "label": "unconstrained", "color": "tab:blue"},
)
PartialDependenceDisplay.from_estimator(
rf_cst,
X,
features=[0],
line_kw={"linewidth": 4, "label": "constrained", "color": "tab:orange"},
ax=disp.axes_,
)
disp.axes_[0, 0].plot(
X[:, 0], y, "o", alpha=0.5, zorder=-1, label="samples", color="tab:green"
)
disp.axes_[0, 0].set_ylim(-3, 3)
disp.axes_[0, 0].set_xlim(-1, 1)
disp.axes_[0, 0].legend()
plt.show()
تحسين عرض المحلل#
تم تحسين عرض المحلل: إذا نظرنا إلى forest، المحدد أعلاه:
forest
يمكن الوصول إلى وثائق المحلل بالنقر على أيقونة "؟" في الزاوية اليمنى العليا من المخطط.
بالإضافة إلى ذلك، يتغير لون العرض من البرتقالي إلى الأزرق، عندما يتم تدريب المحلل. يمكنك أيضًا الحصول على هذه المعلومات من خلال التمرير فوق أيقونة "i".
from sklearn.base import clone
clone(forest) # النسخة ليست مدربة
دعم توجيه البيانات الوصفية#
يدعم العديد من المحللين الفائقين وروتينات التحقق المتقاطع الآن توجيه البيانات الوصفية، والتي يتم سردها في دليل المستخدم. على سبيل المثال، هذه هي الطريقة التي يمكنك بها إجراء التحقق المتقاطع المتداخل
مع أوزان العينات و GroupKFold:
import sklearn
from sklearn.metrics import get_scorer
from sklearn.datasets import make_regression
from sklearn.linear_model import Lasso
from sklearn.model_selection import GridSearchCV, cross_validate, GroupKFold
# حاليًا، يتم تعطيل توجيه البيانات الوصفية بشكل افتراضي، ويجب تمكينه بشكل صريح.
sklearn.set_config(enable_metadata_routing=True)
n_samples = 100
X, y = make_regression(n_samples=n_samples, n_features=5, noise=0.5)
rng = np.random.RandomState(7)
groups = rng.randint(0, 10, size=n_samples)
sample_weights = rng.rand(n_samples)
estimator = Lasso().set_fit_request(sample_weight=True)
hyperparameter_grid = {"alpha": [0.1, 0.5, 1.0, 2.0]}
scoring_inner_cv = get_scorer("neg_mean_squared_error").set_score_request(
sample_weight=True
)
inner_cv = GroupKFold(n_splits=5)
grid_search = GridSearchCV(
estimator=estimator,
param_grid=hyperparameter_grid,
cv=inner_cv,
scoring=scoring_inner_cv,
)
outer_cv = GroupKFold(n_splits=5)
scorers = {
"mse": get_scorer("neg_mean_squared_error").set_score_request(sample_weight=True)
}
results = cross_validate(
grid_search,
X,
y,
cv=outer_cv,
scoring=scorers,
return_estimator=True,
params={"sample_weight": sample_weights, "groups": groups},
)
print("cv error on test sets:", results["test_mse"])
# تعيين العلم إلى القيمة الافتراضية `False` لتجنب التداخل مع النصوص الأخرى.
sklearn.set_config(enable_metadata_routing=False)
cv error on test sets: [-0.23753287 -0.33144764 -0.24428617 -0.44702015 -0.53005986]
تحسين كفاءة الذاكرة والوقت لـ PCA على البيانات المتناثرة#
يمكن لـ PCA الآن التعامل مع المصفوفات المتناثرة بشكل أصلي لمحلل arpack من خلال الاستفادة من scipy.sparse.linalg.LinearOperator لتجنب
تجسيد المصفوفات المتناثرة الكبيرة عند إجراء
تحليل القيمة الذاتية لمصفوفة مجموعة البيانات.
from sklearn.decomposition import PCA
import scipy.sparse as sp
from time import time
X_sparse = sp.random(m=1000, n=1000, random_state=0)
X_dense = X_sparse.toarray()
t0 = time()
PCA(n_components=10, svd_solver="arpack").fit(X_sparse)
time_sparse = time() - t0
t0 = time()
PCA(n_components=10, svd_solver="arpack").fit(X_dense)
time_dense = time() - t0
print(f"Speedup: {time_dense / time_sparse:.1f}x")
Speedup: 7.0x
Total running time of the script: (0 minutes 4.779 seconds)
Related examples