ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
مقارنة خوارزميات التجميع K-Means و MiniBatchKMeans#
نريد مقارنة أداء خوارزميتي MiniBatchKMeans و KMeans: خوارزمية MiniBatchKMeans أسرع، لكنها تعطي نتائج مختلفة قليلاً (انظر: Mini Batch K-Means).
سنقوم بتجميع مجموعة من البيانات، أولاً باستخدام KMeans ثم باستخدام MiniBatchKMeans، ورسم النتائج. كما سنقوم برسم النقاط التي تحمل تسميات مختلفة بين الخوارزميتين.
# المؤلفون: مطوّرو scikit-learn
# معرف الترخيص: BSD-3-Clause
توليد البيانات#
نبدأ بتوليد مجموعات البيانات التي سيتم تجميعها.
import matplotlib.pyplot as plt
from sklearn.metrics.pairwise import pairwise_distances_argmin
from sklearn.cluster import MiniBatchKMeans
from sklearn.cluster import KMeans
import time
import numpy as np
from sklearn.datasets import make_blobs
np.random.seed(0)
batch_size = 45
centers = [[1, 1], [-1, -1], [1, -1]]
n_clusters = len(centers)
X, labels_true = make_blobs(n_samples=3000, centers=centers, cluster_std=0.7)
حساب التجميع باستخدام KMeans#
حساب التجميع باستخدام MiniBatchKMeans#
mbk = MiniBatchKMeans(
init="k-means++",
n_clusters=3,
batch_size=batch_size,
n_init=10,
max_no_improvement=10,
verbose=0,
)
t0 = time.time()
mbk.fit(X)
t_mini_batch = time.time() - t0
تحقيق التكافؤ بين المجموعات#
نريد أن يكون نفس اللون للمجموعة نفسها من خوارزميتي MiniBatchKMeans و KMeans. دعنا نوائم مراكز المجموعات حسب الأقرب.
k_means_cluster_centers = k_means.cluster_centers_
order = pairwise_distances_argmin(
k_means.cluster_centers_, mbk.cluster_centers_)
mbk_means_cluster_centers = mbk.cluster_centers_[order]
k_means_labels = pairwise_distances_argmin(X, k_means_cluster_centers)
mbk_means_labels = pairwise_distances_argmin(X, mbk_means_cluster_centers)
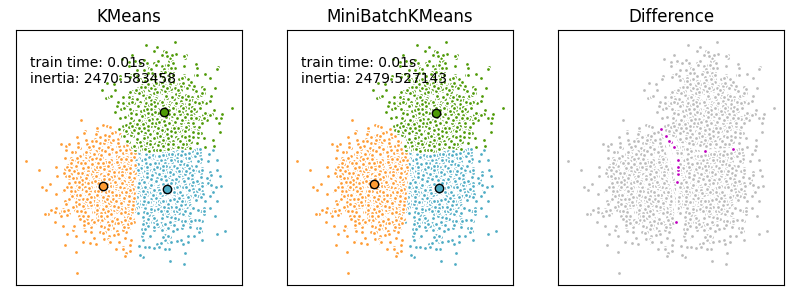
رسم النتائج#
fig = plt.figure(figsize=(8, 3))
fig.subplots_adjust(left=0.02, right=0.98, bottom=0.05, top=0.9)
colors = ["#4EACC5", "#FF9C34", "#4E9A06"]
# KMeans
ax = fig.add_subplot(1, 3, 1)
for k, col in zip(range(n_clusters), colors):
my_members = k_means_labels == k
cluster_center = k_means_cluster_centers[k]
ax.plot(X[my_members, 0], X[my_members, 1],
"w", markerfacecolor=col, marker=".")
ax.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=6,
)
ax.set_title("KMeans")
ax.set_xticks(())
ax.set_yticks(())
plt.text(-3.5, 1.8, "train time: %.2fs\ninertia: %f" %
(t_batch, k_means.inertia_))
# MiniBatchKMeans
ax = fig.add_subplot(1, 3, 2)
for k, col in zip(range(n_clusters), colors):
my_members = mbk_means_labels == k
cluster_center = mbk_means_cluster_centers[k]
ax.plot(X[my_members, 0], X[my_members, 1],
"w", markerfacecolor=col, marker=".")
ax.plot(
cluster_center[0],
cluster_center[1],
"o",
markerfacecolor=col,
markeredgecolor="k",
markersize=6,
)
ax.set_title("MiniBatchKMeans")
ax.set_xticks(())
ax.set_yticks(())
plt.text(-3.5, 1.8, "train time: %.2fs\ninertia: %f" %
(t_mini_batch, mbk.inertia_))
# تهيئة المصفوفة المختلفة إلى كلها False
different = mbk_means_labels == 4
ax = fig.add_subplot(1, 3, 3)
for k in range(n_clusters):
different += (k_means_labels == k) != (mbk_means_labels == k)
identical = np.logical_not(different)
ax.plot(X[identical, 0], X[identical, 1], "w",
markerfacecolor="#bbbbbb", marker=".")
ax.plot(X[different, 0], X[different, 1], "w", markerfacecolor="m", marker=".")
ax.set_title("Difference")
ax.set_xticks(())
ax.set_yticks(())
plt.show()
Total running time of the script: (0 minutes 0.180 seconds)
Related examples