ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
تقييم خوارزميات كشف الشواذ#
يقارن هذا المثال بين خوارزميتين لكشف الشواذ، وهما
مُعامل القيم المتطرفة المحلي (LOF) و غابة العزل (IForest)، على
مجموعات بيانات واقعية متوفرة في sklearn.datasets. الهدف هو إظهار
أن خوارزميات مختلفة تؤدي أداءً جيدًا على مجموعات بيانات مختلفة وتوضيح سرعة
تدريبها وحساسيتها لضبط المعلمات.
تتم تدريب الخوارزميات (بدون تسميات) على مجموعة البيانات الكاملة المفترضة أنها تحتوي على شواذ.
1. يتم حساب منحنيات ROC باستخدام معرفة التسميات الحقيقية
وعرضها باستخدام RocCurveDisplay.
يتم تقييم الأداء من حيث ROC-AUC.
# المؤلفون: مطوري scikit-learn
# معرف SPDX-License: BSD-3-Clause
معالجة البيانات وتدريب النموذج#
تتطلب نماذج كشف الشواذ المختلفة معالجة مسبقة مختلفة. في
وجود متغيرات فئوية،
OrdinalEncoder هي استراتيجية جيدة غالبًا
للنماذج القائمة على الأشجار مثل IsolationForest،
في حين أن النماذج القائمة على الجيران مثل
LocalOutlierFactor
ستتأثر بالترتيب الناتج عن الترميز الترتيبي. لتجنب
فرض ترتيب، يجب استخدام
OneHotEncoder.
قد تتطلب النماذج القائمة على الجيران أيضًا معايرة الميزات الرقمية (انظر
على سبيل المثال تأثير إعادة المعايرة على نماذج k-neighbors). في وجود شواذ، يعد
RobustScaler
خيارًا جيدًا.
from sklearn.compose import ColumnTransformer
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import (
OneHotEncoder,
OrdinalEncoder,
RobustScaler,
)
def make_estimator(name, categorical_columns=None, iforest_kw=None, lof_kw=None):
"""إنشاء نموذج كشف شواذ بناءً على اسمه."""
if name == "LOF":
outlier_detector = LocalOutlierFactor(**(lof_kw or {}))
if categorical_columns is None:
preprocessor = RobustScaler()
else:
preprocessor = ColumnTransformer(
transformers=[("categorical", OneHotEncoder(), categorical_columns)],
remainder=RobustScaler(),
)
else: # name == "IForest"
outlier_detector = IsolationForest(**(iforest_kw or {}))
if categorical_columns is None:
preprocessor = None
else:
ordinal_encoder = OrdinalEncoder(
handle_unknown="use_encoded_value", unknown_value=-1
)
preprocessor = ColumnTransformer(
transformers=[
("categorical", ordinal_encoder, categorical_columns),
],
remainder="passthrough",
)
return make_pipeline(preprocessor, outlier_detector)
تقوم دالة fit_predict التالية بإرجاع متوسط درجة الشذوذ لـ X.
from time import perf_counter
def fit_predict(estimator, X):
tic = perf_counter()
if estimator[-1].__class__.__name__ == "LocalOutlierFactor":
estimator.fit(X)
y_pred = estimator[-1].negative_outlier_factor_
else: # "IsolationForest"
y_pred = estimator.fit(X).decision_function(X)
toc = perf_counter()
print(f"Duration for {model_name}: {toc - tic:.2f} s")
return y_pred
في بقية المثال، نقوم بمعالجة مجموعة بيانات واحدة لكل قسم. بعد تحميل
البيانات، يتم تعديل الأهداف لتشمل فئتين: 0 تمثل
القيم العادية و 1 تمثل الشواذ. بسبب القيود الحسابية لوثائق scikit-learn،
يتم تقليل حجم العينة لبعض مجموعات البيانات باستخدام
train_test_split.
علاوة على ذلك، نقوم بضبط n_neighbors ليتناسب مع العدد المتوقع للشواذ
expected_n_anomalies = n_samples * expected_anomaly_fraction. هذه هي
استراتيجية جيدة طالما أن نسبة الشواذ ليست منخفضة جدًا، والسبب
هو أن n_neighbors يجب أن يكون أكبر من عدد العينات
في المجموعة الأقل كثافة (انظر
الكشف عن القيم الشاذة باستخدام عامل الانحراف المحلي (LOF)).
KDDCup99 - مجموعة بيانات SA#
تم إنشاء مجموعة بيانات Kddcup 99 dataset باستخدام شبكة مغلقة وهجمات محقونة يدويًا. مجموعة بيانات SA هي جزء فرعي منها تم الحصول عليه ببساطة عن طريق اختيار جميع البيانات العادية ونسبة شواذ تبلغ حوالي 3%.
import numpy as np
from sklearn.datasets import fetch_kddcup99
from sklearn.model_selection import train_test_split
X, y = fetch_kddcup99(
subset="SA", percent10=True, random_state=42, return_X_y=True, as_frame=True
)
y = (y != b"normal.").astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.1, stratify=y, random_state=42)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
10065 datapoints with 338 anomalies (3.36%)
تحتوي مجموعة بيانات SA على 41 ميزة، منها 3 فئوية: "protocol_type"، "service" و "flag".
y_true = {}
y_pred = {"LOF": {}, "IForest": {}}
model_names = ["LOF", "IForest"]
cat_columns = ["protocol_type", "service", "flag"]
y_true["KDDCup99 - SA"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["KDDCup99 - SA"] = fit_predict(model, X)
Duration for LOF: 1.35 s
Duration for IForest: 0.43 s
مجموعة بيانات غطاء الغابات#
مجموعة بيانات Forest covertypes هي مجموعة بيانات متعددة الفئات حيث الهدف هو النوع السائد من الأشجار في رقعة معينة من الغابة. تحتوي على 54 ميزة، بعضها ("Wilderness_Area" و "Soil_Type") مشفرة بالفعل بشكل ثنائي. على الرغم من أنها كانت في الأصل مهمة تصنيف، يمكن اعتبار القيم العادية كعينات مشفرة بالعلامة 2 والشواذ كعينات مشفرة بالعلامة 4.
from sklearn.datasets import fetch_covtype
X, y = fetch_covtype(return_X_y=True, as_frame=True)
s = (y == 2) + (y == 4)
X = X.loc[s]
y = y.loc[s]
y = (y != 2).astype(np.int32)
X, _, y, _ = train_test_split(X, y, train_size=0.05, stratify=y, random_state=42)
X_forestcover = X # save X for later use
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
14302 datapoints with 137 anomalies (0.96%)
y_true["forestcover"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["forestcover"] = fit_predict(model, X)
Duration for LOF: 1.46 s
Duration for IForest: 0.26 s
مجموعة بيانات Ames Housing#
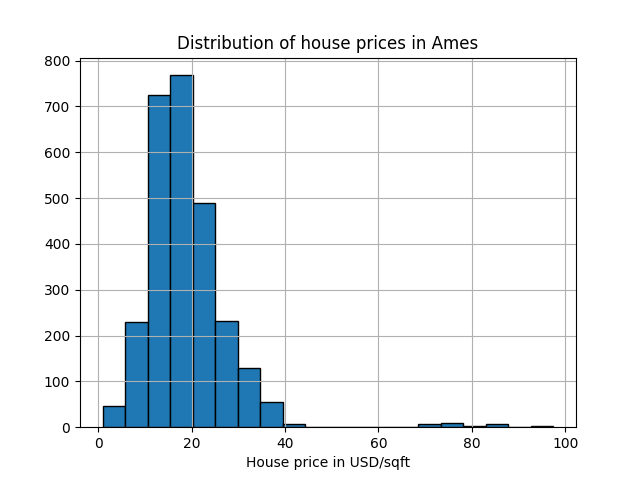
مجموعة بيانات Ames housing dataset <http://www.openml.org/d/43926>`_ هي في الأصل مجموعة بيانات للتنبؤ حيث الهدف هو أسعار بيع المنازل في Ames، أيوا. هنا نحولها إلى مشكلة كشف شواذ من خلال اعتبار المنازل التي يزيد سعرها عن 70 دولار/قدم مربع كشواذ. لجعل المشكلة أسهل، نقوم بإسقاط الأسعار المتوسطة بين 40 و 70 دولار/قدم مربع.
import matplotlib.pyplot as plt
from sklearn.datasets import fetch_openml
X, y = fetch_openml(name="ames_housing", version=1, return_X_y=True, as_frame=True)
y = y.div(X["Lot_Area"])
# None values in pandas 1.5.1 were mapped to np.nan in pandas 2.0.1
X["Misc_Feature"] = X["Misc_Feature"].cat.add_categories("NoInfo").fillna("NoInfo")
X["Mas_Vnr_Type"] = X["Mas_Vnr_Type"].cat.add_categories("NoInfo").fillna("NoInfo")
X.drop(columns="Lot_Area", inplace=True)
mask = (y < 40) | (y > 70)
X = X.loc[mask]
y = y.loc[mask]
y.hist(bins=20, edgecolor="black")
plt.xlabel("House price in USD/sqft")
_ = plt.title("Distribution of house prices in Ames")
y = (y > 70).astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2714 datapoints with 30 anomalies (1.11%)
تحتوي مجموعة البيانات على 46 ميزة فئوية. في هذه الحالة، من الأسهل استخدام
make_column_selector للعثور عليها بدلاً من تمرير
قائمة تم إنشاؤها يدويًا.
from sklearn.compose import make_column_selector as selector
categorical_columns_selector = selector(dtype_include="category")
cat_columns = categorical_columns_selector(X)
y_true["ames_housing"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
categorical_columns=cat_columns,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["ames_housing"] = fit_predict(model, X)
Duration for LOF: 0.86 s
Duration for IForest: 0.27 s
مجموعة بيانات Cardiotocography#
مجموعة بيانات Cardiotocography dataset <http://www.openml.org/d/1466>`_ هي مجموعة بيانات متعددة الفئات من تخطيط القلب للجنين، حيث الفئات هي نمط معدل ضربات القلب الجنيني (FHR) مشفرة بعلامات من 1 إلى 10. هنا نحدد الفئة 3 (الفئة الأقلية) لتمثل الشواذ. تحتوي على 30 ميزة رقمية، بعضها مشفر بشكل ثنائي وبعضها مستمر.
X, y = fetch_openml(name="cardiotocography", version=1, return_X_y=True, as_frame=False)
X_cardiotocography = X # save X for later use
s = y == "3"
y = s.astype(np.int32)
n_samples, anomaly_frac = X.shape[0], y.mean()
print(f"{n_samples} datapoints with {y.sum()} anomalies ({anomaly_frac:.02%})")
2126 datapoints with 53 anomalies (2.49%)
y_true["cardiotocography"] = y
for model_name in model_names:
model = make_estimator(
name=model_name,
lof_kw={"n_neighbors": int(n_samples * anomaly_frac)},
iforest_kw={"random_state": 42},
)
y_pred[model_name]["cardiotocography"] = fit_predict(model, X)
Duration for LOF: 0.04 s
Duration for IForest: 0.17 s
رسم وتفسير النتائج#
يرتبط أداء الخوارزمية بمدى جودة معدل الإيجابيات الحقيقية (TPR) عند قيم منخفضة لمعدل الإيجابيات الخاطئة (FPR). أفضل الخوارزميات لديها المنحنى في أعلى يسار الرسم البياني ومساحة تحت المنحنى (AUC) قريبة من 1. الخط المتقطع المائل يمثل تصنيفًا عشوائيًا للشواذ والقيم العادية.
import math
from sklearn.metrics import RocCurveDisplay
cols = 2
pos_label = 0 # mean 0 belongs to positive class
datasets_names = y_true.keys()
rows = math.ceil(len(datasets_names) / cols)
fig, axs = plt.subplots(nrows=rows, ncols=cols, squeeze=False, figsize=(10, rows * 4))
for ax, dataset_name in zip(axs.ravel(), datasets_names):
for model_idx, model_name in enumerate(model_names):
display = RocCurveDisplay.from_predictions(
y_true[dataset_name],
y_pred[model_name][dataset_name],
pos_label=pos_label,
name=model_name,
ax=ax,
plot_chance_level=(model_idx == len(model_names) - 1),
chance_level_kw={"linestyle": ":"},
)
ax.set_title(dataset_name)
_ = plt.tight_layout(pad=2.0) # spacing between subplots
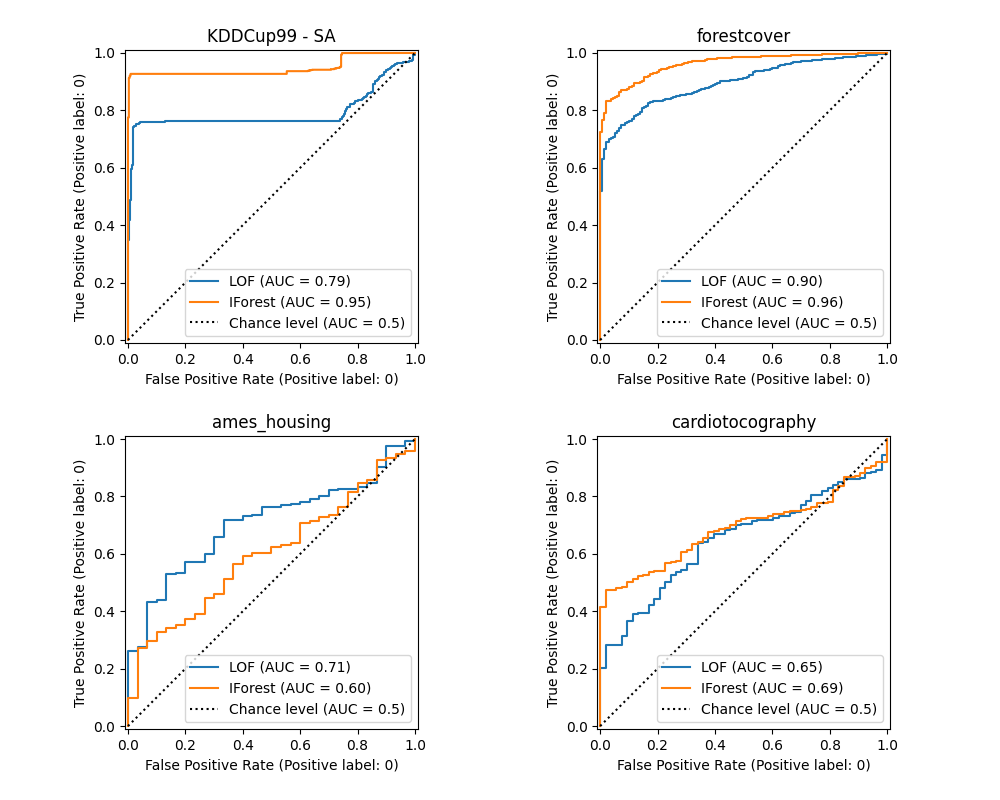
نلاحظ أنه بمجرد ضبط عدد الجيران، يؤدي LOF و IForest أداءً مشابهًا من حيث ROC AUC لمجموعات بيانات forestcover و cardiotocography. النتيجة أفضل قليلاً لـ IForest في مجموعة بيانات SA، بينما يؤدي LOF أداءً أفضل بكثير في مجموعة بيانات Ames housing مقارنة بـ IForest.
تذكر مع ذلك أن Isolation Forest تميل إلى التدريب بشكل أسرع من LOF على مجموعات البيانات ذات العدد الكبير من العينات. يحتاج LOF إلى حساب المسافات الزوجية لإيجاد الجيران الأقرب، مما له تعقيد تربيعي فيما يتعلق بعدد الملاحظات. يمكن أن يجعل هذا الأسلوب محظورًا على مجموعات البيانات الكبيرة.
دراسة التخفيف#
في هذا القسم، نستكشف تأثير المعلمة n_neighbors
واختيار معايرة الميزات الرقمية على نموذج LOF. هنا نستخدم
Forest covertypes حيث يتم إدخال الفئات المشفرة بشكل ثنائي
مقياسًا طبيعيًا للمسافات الإقليدية بين 0 و 1. بعد ذلك، نريد طريقة
للمعايرة لتجنب منح امتياز للميزات غير الثنائية وأن تكون قوية بما يكفي
للشواذ بحيث لا تصبح مهمة العثور عليها صعبة للغاية.
X = X_forestcover
y = y_true["forestcover"]
n_samples = X.shape[0]
n_neighbors_list = (n_samples * np.array([0.2, 0.02, 0.01, 0.001])).astype(np.int32)
model = make_pipeline(RobustScaler(), LocalOutlierFactor())
linestyles = ["solid", "dashed", "dashdot", ":", (5, (10, 3))]
fig, ax = plt.subplots()
for model_idx, (linestyle, n_neighbors) in enumerate(zip(linestyles, n_neighbors_list)):
model.set_params(localoutlierfactor__n_neighbors=n_neighbors)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=f"n_neighbors = {n_neighbors}",
ax=ax,
plot_chance_level=(model_idx == len(n_neighbors_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("RobustScaler with varying n_neighbors\non forestcover dataset")
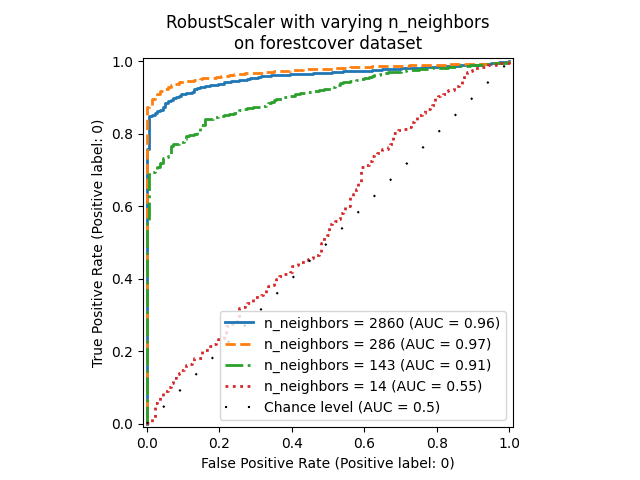
نلاحظ أن عدد الجيران له تأثير كبير على أداء
النموذج. إذا كان لدى المرء إمكانية الوصول إلى (بعض على الأقل) تصنيفات أرضية حقيقية،
فمن المهم عندئذٍ ضبط n_neighbors وفقًا لذلك. تتمثل إحدى الطرق الملائمة للقيام بذلك
في استكشاف قيم n_neighbors من حيث الحجم المتوقع
للتلوث.
from sklearn.preprocessing import MinMaxScaler, SplineTransformer, StandardScaler
preprocessor_list = [
None,
RobustScaler(),
StandardScaler(),
MinMaxScaler(),
SplineTransformer(),
]
expected_anomaly_fraction = 0.02
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
_ = ax.set_title("n_neighbors ثابت مع معالجة مسبقة متغيرة\nعلى مجموعة بيانات forestcover")
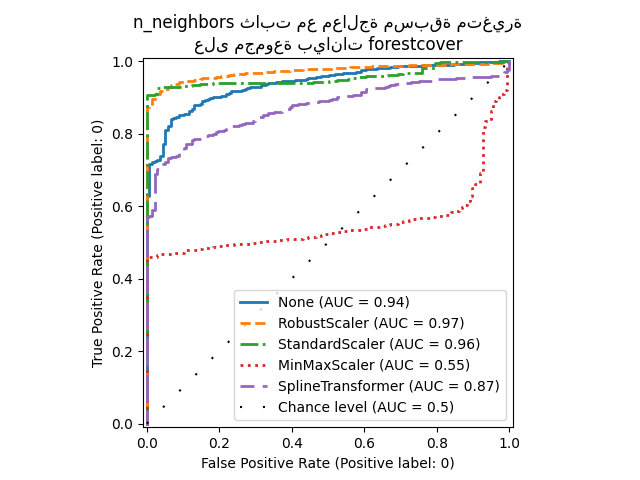
من ناحية، يقوم RobustScaler بقياس
كل ميزة بشكل مستقل باستخدام النطاق الربيعي (IQR) افتراضيًا، وهو
النطاق بين النسبتين المئويتين 25 و 75 من البيانات. يقوم بتركيز
البيانات عن طريق طرح الوسيط ثم قياسها بالقسمة على IQR. IQR
مقاوم للقيم المتطرفة: يتأثر الوسيط والنطاق الربيعي بالقيم
المتطرفة بدرجة أقل من النطاق والمتوسط والانحراف
المعياري. علاوة على ذلك، لا يقوم RobustScaler
بسحق القيم المتطرفة الهامشية، على عكس
StandardScaler.
من ناحية أخرى، يقوم MinMaxScaler بقياس
كل ميزة بشكل فردي بحيث يتم تعيين نطاقها في النطاق بين الصفر
والواحد. إذا كانت هناك قيم متطرفة في البيانات، فيمكنها تحريفها نحو
الحد الأدنى أو الحد الأقصى للقيم، مما يؤدي إلى توزيع مختلف تمامًا
للبيانات مع قيم متطرفة هامشية كبيرة: يمكن طي جميع القيم غير المتطرفة
تقريبًا معًا نتيجة لذلك.
قمنا أيضًا بتقييم عدم وجود معالجة مسبقة على الإطلاق (عن طريق تمرير None
إلى خط الأنابيب)، و StandardScaler و
SplineTransformer. يرجى الرجوع إلى
وثائق كل منها لمزيد من التفاصيل.
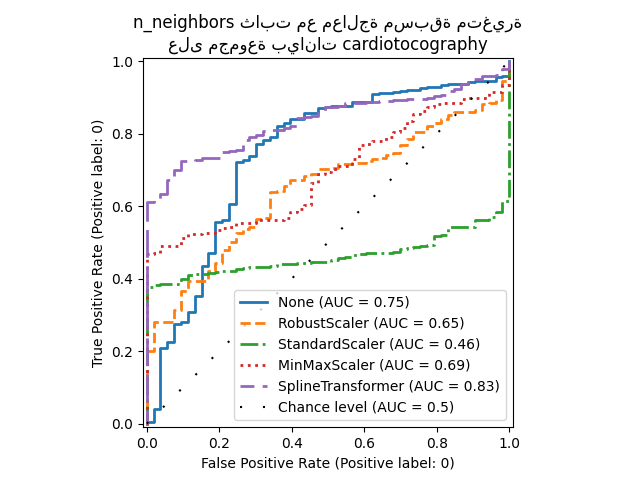
لاحظ أن المعالجة المسبقة المثلى تعتمد على مجموعة البيانات، كما هو موضح أدناه:
X = X_cardiotocography
y = y_true["cardiotocography"]
n_samples, expected_anomaly_fraction = X.shape[0], 0.025
lof = LocalOutlierFactor(n_neighbors=int(n_samples * expected_anomaly_fraction))
fig, ax = plt.subplots()
for model_idx, (linestyle, preprocessor) in enumerate(
zip(linestyles, preprocessor_list)
):
model = make_pipeline(preprocessor, lof)
model.fit(X)
y_pred = model[-1].negative_outlier_factor_
display = RocCurveDisplay.from_predictions(
y,
y_pred,
pos_label=pos_label,
name=str(preprocessor).split("(")[0],
ax=ax,
plot_chance_level=(model_idx == len(preprocessor_list) - 1),
chance_level_kw={"linestyle": (0, (1, 10))},
linestyle=linestyle,
linewidth=2,
)
ax.set_title(
"n_neighbors ثابت مع معالجة مسبقة متغيرة\nعلى مجموعة بيانات cardiotocography"
)
plt.show()
Total running time of the script: (0 minutes 48.741 seconds)
Related examples
الكشف عن القيم الشاذة باستخدام عامل الانحراف المحلي (LOF)
مقارنة خوارزميات الكشف عن الشذوذ لكشف القيم المتطرفة في مجموعات بيانات تجريبية
مقارنة تأثير المقياس المختلف على البيانات مع القيم الشاذة