ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
استنتاج القيم المفقودة قبل بناء أداة تقدير#
يمكن استبدال القيم المفقودة بالمتوسط أو الوسيط أو القيمة الأكثر تكرارًا باستخدام SimpleImputer الأساسي.
في هذا المثال، سنبحث في تقنيات الاستنتاج المختلفة:
الاستنتاج بالقيمة الثابتة 0
الاستنتاج بواسطة القيمة المتوسطة لكل ميزة مدمجة مع متغير مساعد لمؤشر الفقد
استنتاج أقرب جار k
الاستنتاج التكراري
سنستخدم مجموعتي بيانات: مجموعة بيانات مرض السكري التي تتكون من 10 متغيرات ميزات تم جمعها من مرضى السكري بهدف التنبؤ بتطور المرض ومجموعة بيانات إسكان كاليفورنيا التي يكون هدفها متوسط قيمة المنزل لمقاطعات كاليفورنيا.
نظرًا لعدم وجود قيم مفقودة في أي من مجموعتي البيانات هذه، فسنزيل بعض القيم لإنشاء إصدارات جديدة ببيانات مفقودة بشكل مصطنع. تتم بعد ذلك مقارنة أداء RandomForestRegressor على مجموعة البيانات الأصلية الكاملة بالأداء على مجموعات البيانات المعدلة مع القيم المفقودة بشكل مصطنع المستنتجة باستخدام تقنيات مختلفة.
# Authors: The scikit-learn developers
# SPDX-License-Identifier: BSD-3-Clause
تنزيل البيانات وإنشاء مجموعات القيم المفقودة#
أولاً، نقوم بتنزيل مجموعتي البيانات. يتم شحن مجموعة بيانات مرض السكري مع scikit-learn. تحتوي على 442 إدخالًا، لكل منها 10 ميزات. مجموعة بيانات إسكان كاليفورنيا أكبر بكثير مع 20640 إدخالًا و 8 ميزات. يجب تنزيلها. سنستخدم فقط أول 400 إدخال لتسريع العمليات الحسابية ولكن لا تتردد في استخدام مجموعة البيانات بأكملها.
import numpy as np
from sklearn.datasets import fetch_california_housing, load_diabetes
rng = np.random.RandomState(42)
X_diabetes, y_diabetes = load_diabetes(return_X_y=True)
X_california, y_california = fetch_california_housing(return_X_y=True)
X_california = X_california[:300]
y_california = y_california[:300]
X_diabetes = X_diabetes[:300]
y_diabetes = y_diabetes[:300]
def add_missing_values(X_full, y_full):
n_samples, n_features = X_full.shape
# إضافة قيم مفقودة في 75٪ من الأسطر
missing_rate = 0.75
n_missing_samples = int(n_samples * missing_rate)
missing_samples = np.zeros(n_samples, dtype=bool)
missing_samples[:n_missing_samples] = True
rng.shuffle(missing_samples)
missing_features = rng.randint(0, n_features, n_missing_samples)
X_missing = X_full.copy()
X_missing[missing_samples, missing_features] = np.nan
y_missing = y_full.copy()
return X_missing, y_missing
X_miss_california, y_miss_california = add_missing_values(X_california, y_california)
X_miss_diabetes, y_miss_diabetes = add_missing_values(X_diabetes, y_diabetes)
استنتاج البيانات المفقودة وتسجيلها#
الآن سنكتب دالة ستسجل النتائج على البيانات المستنتجة بشكل مختلف. دعونا نلقي نظرة على كل أداة استنتاج على حدة:
rng = np.random.RandomState(0)
from sklearn.ensemble import RandomForestRegressor
# لاستخدام IterativeImputer التجريبي، نحتاج إلى طلبه صراحةً:
from sklearn.experimental import enable_iterative_imputer # noqa
from sklearn.impute import IterativeImputer, KNNImputer, SimpleImputer
from sklearn.model_selection import cross_val_score
from sklearn.pipeline import make_pipeline
N_SPLITS = 4
regressor = RandomForestRegressor(random_state=0)
المعلومات المفقودة#
بالإضافة إلى استنتاج القيم المفقودة، تحتوي أدوات الاستنتاج على معامل add_indicator يضع علامة على القيم المفقودة، والتي قد تحمل بعض المعلومات.
def get_scores_for_imputer(imputer, X_missing, y_missing):
estimator = make_pipeline(imputer, regressor)
impute_scores = cross_val_score(
estimator, X_missing, y_missing, scoring="neg_mean_squared_error", cv=N_SPLITS
)
return impute_scores
x_labels = []
mses_california = np.zeros(5)
stds_california = np.zeros(5)
mses_diabetes = np.zeros(5)
stds_diabetes = np.zeros(5)
تقدير النتيجة#
أولاً، نريد تقدير النتيجة على البيانات الأصلية:
def get_full_score(X_full, y_full):
full_scores = cross_val_score(
regressor, X_full, y_full, scoring="neg_mean_squared_error", cv=N_SPLITS
)
return full_scores.mean(), full_scores.std()
mses_california[0], stds_california[0] = get_full_score(X_california, y_california)
mses_diabetes[0], stds_diabetes[0] = get_full_score(X_diabetes, y_diabetes)
x_labels.append("البيانات الكاملة")
استبدال القيم المفقودة بـ 0#
الآن سنقوم بتقدير النتيجة على البيانات حيث يتم استبدال القيم المفقودة بـ 0:
def get_impute_zero_score(X_missing, y_missing):
imputer = SimpleImputer(
missing_values=np.nan, add_indicator=True, strategy="constant", fill_value=0
)
zero_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return zero_impute_scores.mean(), zero_impute_scores.std()
mses_california[1], stds_california[1] = get_impute_zero_score(
X_miss_california, y_miss_california
)
mses_diabetes[1], stds_diabetes[1] = get_impute_zero_score(
X_miss_diabetes, y_miss_diabetes
)
x_labels.append("استنتاج الصفر")
استنتاج kNN للقيم المفقودة#
KNNImputer يستنتج القيم المفقودة باستخدام المتوسط المرجح أو غير المرجح للعدد المطلوب من أقرب الجيران.
def get_impute_knn_score(X_missing, y_missing):
imputer = KNNImputer(missing_values=np.nan, add_indicator=True)
knn_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return knn_impute_scores.mean(), knn_impute_scores.std()
mses_california[2], stds_california[2] = get_impute_knn_score(
X_miss_california, y_miss_california
)
mses_diabetes[2], stds_diabetes[2] = get_impute_knn_score(
X_miss_diabetes, y_miss_diabetes
)
x_labels.append("استنتاج KNN")
استنتاج القيم المفقودة بالمتوسط#
def get_impute_mean(X_missing, y_missing):
imputer = SimpleImputer(missing_values=np.nan, strategy="mean", add_indicator=True)
mean_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return mean_impute_scores.mean(), mean_impute_scores.std()
mses_california[3], stds_california[3] = get_impute_mean(
X_miss_california, y_miss_california
)
mses_diabetes[3], stds_diabetes[3] = get_impute_mean(X_miss_diabetes, y_miss_diabetes)
x_labels.append("استنتاج المتوسط")
الاستنتاج التكراري للقيم المفقودة#
خيار آخر هو IterativeImputer. يستخدم هذا الانحدار الخطي الدائري، نمذجة كل ميزة ذات قيم مفقودة كدالة لميزات أخرى، بدورها.
يفترض الإصدار المطبق متغيرات غاوسية (ناتجة). إذا كانت ميزاتك غير طبيعية بشكل واضح، ففكر في تحويلها لتبدو أكثر طبيعية لتحسين الأداء المحتمل.
def get_impute_iterative(X_missing, y_missing):
imputer = IterativeImputer(
missing_values=np.nan,
add_indicator=True,
random_state=0,
n_nearest_features=3,
max_iter=1,
sample_posterior=True,
)
iterative_impute_scores = get_scores_for_imputer(imputer, X_missing, y_missing)
return iterative_impute_scores.mean(), iterative_impute_scores.std()
mses_california[4], stds_california[4] = get_impute_iterative(
X_miss_california, y_miss_california
)
mses_diabetes[4], stds_diabetes[4] = get_impute_iterative(
X_miss_diabetes, y_miss_diabetes
)
x_labels.append("الاستنتاج التكراري")
mses_diabetes = mses_diabetes * -1
mses_california = mses_california * -1
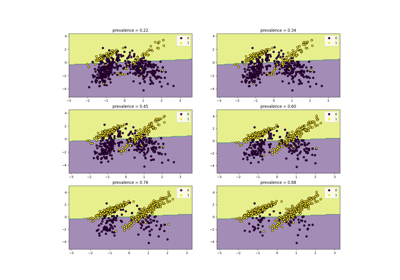
رسم النتائج#
أخيرًا، سنقوم بتصور النتيجة:
import matplotlib.pyplot as plt
n_bars = len(mses_diabetes)
xval = np.arange(n_bars)
colors = ["r", "g", "b", "orange", "black"]
# plot diabetes results
plt.figure(figsize=(12, 6))
ax1 = plt.subplot(121)
for j in xval:
ax1.barh(
j,
mses_diabetes[j],
xerr=stds_diabetes[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax1.set_title("تقنيات الاستنتاج مع بيانات السكري")
ax1.set_xlim(left=np.min(mses_diabetes) * 0.9, right=np.max(mses_diabetes) * 1.1)
ax1.set_yticks(xval)
ax1.set_xlabel("MSE")
ax1.invert_yaxis()
ax1.set_yticklabels(x_labels)
# plot california dataset results
ax2 = plt.subplot(122)
for j in xval:
ax2.barh(
j,
mses_california[j],
xerr=stds_california[j],
color=colors[j],
alpha=0.6,
align="center",
)
ax2.set_title("تقنيات الاستنتاج مع بيانات كاليفورنيا")
ax2.set_yticks(xval)
ax2.set_xlabel("MSE")
ax2.invert_yaxis()
ax2.set_yticklabels([""] * n_bars)
plt.show()
يمكنك أيضًا تجربة تقنيات مختلفة. على سبيل المثال، الوسيط هو مقدر أكثر قوة للبيانات ذات المتغيرات ذات الحجم الكبير والتي يمكن أن تهيمن على النتائج (المعروفة أيضًا باسم "الذيل الطويل").
Total running time of the script: (0 minutes 9.716 seconds)
Related examples
استنتاج القيم المفقودة مع متغيرات من IterativeImputer
تحليل نوع أولوية التركيز لخوارزمية التباين بايزي غاوسي ميكسشر
أبرز الميزات الجديدة في الإصدار 1.5 من scikit-learn