ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
تقريب خريطة الميزات الصريحة لنواة RBF#
مثال يوضح تقريب خريطة الميزات لنواة RBF.
يوضح كيفية استخدام RBFSampler و Nystroem لتقريب
خريطة الميزات لنواة RBF للتصنيف باستخدام SVM على
مجموعة بيانات الأرقام. يتم مقارنة النتائج باستخدام SVM الخطي في المساحة الأصلية، وSVM الخطي
باستخدام الخرائط التقريبية واستخدام SVM المؤكد.
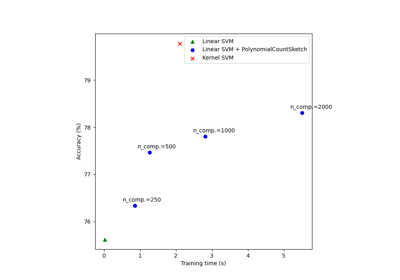
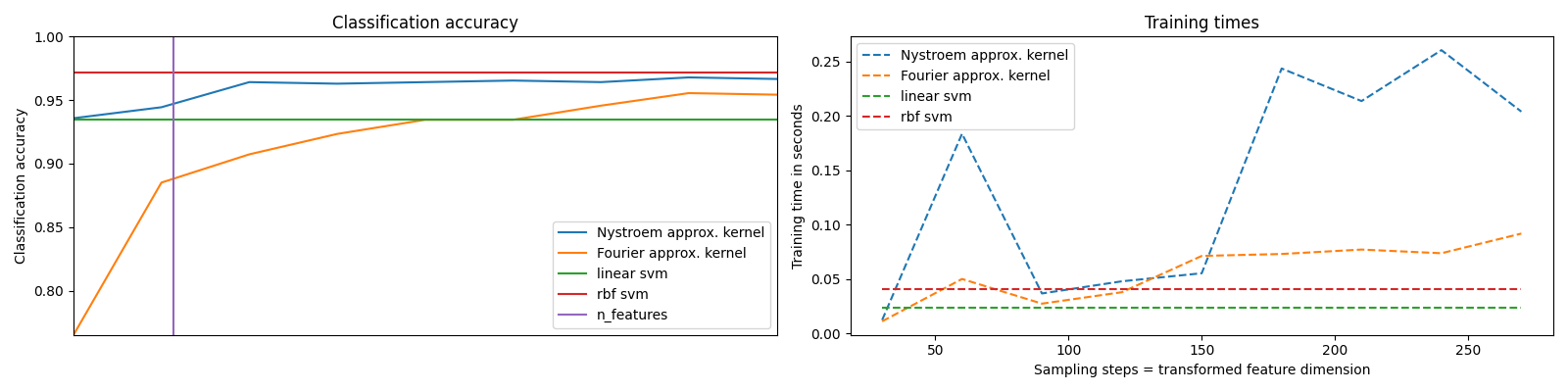
يتم عرض أوقات ودقة لكميات مختلفة من العينات العشوائية (في حالة
RBFSampler، الذي يستخدم ميزات عشوائية فورييه) ومجموعات فرعية مختلفة الحجم
من مجموعة التدريب (لـ Nystroem) لخريطة التقريب.
يرجى ملاحظة أن مجموعة البيانات هنا ليست كبيرة بما يكفي لإظهار فوائد تقريب النواة، حيث أن SVM الدقيق لا يزال سريعًا بشكل معقول.
من الواضح أن أخذ العينات من أبعاد أكثر يؤدي إلى نتائج تصنيف أفضل، ولكن
يأتي بتكلفة أكبر. وهذا يعني أن هناك مفاضلة بين وقت التشغيل
والدقة، والتي يحددها المعامل n_components. لاحظ أن حل SVM الخطي
وأيضًا SVM النواة التقريبية يمكن تسريعه بشكل كبير باستخدام
التدرج العشوائي عبر SGDClassifier.
هذا ليس ممكنًا بسهولة في حالة SVM المؤكد.
# %% # استيرادات حزم وبيانات بايثون، تحميل مجموعة البيانات # ---------------------------------------------------
# المؤلفون: مطوري scikit-learn
# معرف SPDX-License: BSD-3-Clause
# استيرادات بايثون العلمية القياسية
from time import time
import matplotlib.pyplot as plt
import numpy as np
# import مجموعات البيانات، والتصنيفات، ومقاييس الأداء
from sklearn import datasets, pipeline, svm
from sklearn.decomposition import PCA
from sklearn.kernel_approximation import Nystroem, RBFSampler
# مجموعة بيانات الأرقام
digits = datasets.load_digits(n_class=9)
مخططات الوقت والدقة#
لتطبيق مصنف على هذه البيانات، نحتاج إلى تسطيح الصورة، لتحويل البيانات إلى مصفوفة (samples, feature):
n_samples = len(digits.data)
data = digits.data / 16.0
data -= data.mean(axis=0)
# نتعلم الأرقام على النصف الأول من الأرقام
data_train, targets_train = (data[: n_samples // 2], digits.target[: n_samples // 2])
# الآن نتوقع قيمة الرقم على النصف الثاني:
data_test, targets_test = (data[n_samples // 2 :], digits.target[n_samples // 2 :])
# data_test = scaler.transform(data_test)
# إنشاء مصنف: مصنف ناقل الدعم
kernel_svm = svm.SVC(gamma=0.2)
linear_svm = svm.LinearSVC(random_state=42)
# إنشاء خط أنابيب من تقريب النواة
# وSVM الخطي
feature_map_fourier = RBFSampler(gamma=0.2, random_state=1)
feature_map_nystroem = Nystroem(gamma=0.2, random_state=1)
fourier_approx_svm = pipeline.Pipeline(
[
("feature_map", feature_map_fourier),
("svm", svm.LinearSVC(random_state=42)),
]
)
nystroem_approx_svm = pipeline.Pipeline(
[
("feature_map", feature_map_nystroem),
("svm", svm.LinearSVC(random_state=42)),
]
)
# تناسب والتنبؤ باستخدام SVM الخطي والنواة:
kernel_svm_time = time()
kernel_svm.fit(data_train, targets_train)
kernel_svm_score = kernel_svm.score(data_test, targets_test)
kernel_svm_time = time() - kernel_svm_time
linear_svm_time = time()
linear_svm.fit(data_train, targets_train)
linear_svm_score = linear_svm.score(data_test, targets_test)
linear_svm_time = time() - linear_svm_time
sample_sizes = 30 * np.arange(1, 10)
fourier_scores = []
nystroem_scores = []
fourier_times = []
nystroem_times = []
for D in sample_sizes:
fourier_approx_svm.set_params(feature_map__n_components=D)
nystroem_approx_svm.set_params(feature_map__n_components=D)
start = time()
nystroem_approx_svm.fit(data_train, targets_train)
nystroem_times.append(time() - start)
start = time()
fourier_approx_svm.fit(data_train, targets_train)
fourier_times.append(time() - start)
fourier_score = fourier_approx_svm.score(data_test, targets_test)
nystroem_score = nystroem_approx_svm.score(data_test, targets_test)
nystroem_scores.append(nystroem_score)
fourier_scores.append(fourier_score)
# رسم النتائج:
plt.figure(figsize=(16, 4))
accuracy = plt.subplot(121)
# محور y الثاني للأوقات
timescale = plt.subplot(122)
accuracy.plot(sample_sizes, nystroem_scores, label="Nystroem approx. kernel")
timescale.plot(sample_sizes, nystroem_times, "--", label="Nystroem approx. kernel")
accuracy.plot(sample_sizes, fourier_scores, label="Fourier approx. kernel")
timescale.plot(sample_sizes, fourier_times, "--", label="Fourier approx. kernel")
# خطوط أفقية للنواة rbf والنواة الخطية:
accuracy.plot(
[sample_sizes[0], sample_sizes[-1]],
[linear_svm_score, linear_svm_score],
label="linear svm",
)
timescale.plot(
[sample_sizes[0], sample_sizes[-1]],
[linear_svm_time, linear_svm_time],
"--",
label="linear svm",
)
accuracy.plot(
[sample_sizes[0], sample_sizes[-1]],
[kernel_svm_score, kernel_svm_score],
label="rbf svm",
)
timescale.plot(
[sample_sizes[0], sample_sizes[-1]],
[kernel_svm_time, kernel_svm_time],
"--",
label="rbf svm",
)
# خط عمودي لأبعاد مجموعة البيانات = 64
accuracy.plot([64, 64], [0.7, 1], label="n_features")
# الأساطير والعلامات
accuracy.set_title("Classification accuracy")
timescale.set_title("Training times")
accuracy.set_xlim(sample_sizes[0], sample_sizes[-1])
accuracy.set_xticks(())
accuracy.set_ylim(np.min(fourier_scores), 1)
timescale.set_xlabel("Sampling steps = transformed feature dimension")
accuracy.set_ylabel("Classification accuracy")
timescale.set_ylabel("Training time in seconds")
accuracy.legend(loc="best")
timescale.legend(loc="best")
plt.tight_layout()
plt.show()
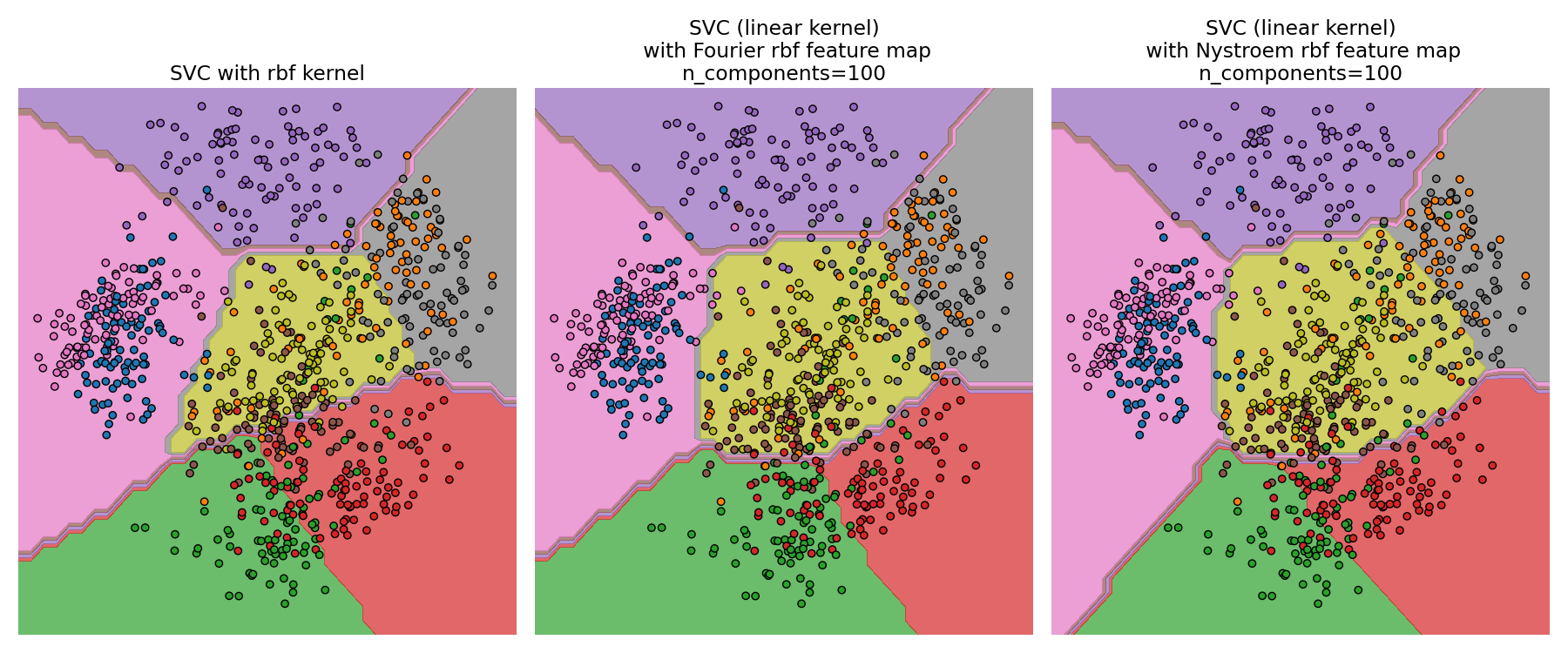
أسطح القرار لSVM النواة RBF وSVM الخطي#
المخطط الثاني يصور أسطح القرار لSVM النواة RBF و
SVM الخطي مع خرائط النواة التقريبية.
يظهر المخطط أسطح القرار للمصنفين المسقطة على
أول مكونين رئيسيين للبيانات. يجب أخذ هذا التصور مع حبة من الملح حيث أنه مجرد شريحة مثيرة للاهتمام
عبر سطح القرار في 64 بُعدًا. لاحظ بشكل خاص أن
نقطة البيانات (الممثلة كنقطة) لا يتم تصنيفها بالضرورة
في المنطقة التي تقع فيها، حيث لن تقع على المستوى
الذي يمتد عبر أول مكونين رئيسيين.
يتم وصف استخدام RBFSampler و Nystroem بالتفصيل
في Kernel Approximation.
# تصور سطح القرار، المسقط إلى أول
# مكونين رئيسيين لمجموعة البيانات
pca = PCA(n_components=8, random_state=42).fit(data_train)
X = pca.transform(data_train)
# إنشاء شبكة على طول أول مكونين رئيسيين
multiples = np.arange(-2, 2, 0.1)
# خطوات على طول المكون الأول
first = multiples[:, np.newaxis] * pca.components_[0, :]
# خطوات على طول المكون الثاني
second = multiples[:, np.newaxis] * pca.components_[1, :]
# الجمع
grid = first[np.newaxis, :, :] + second[:, np.newaxis, :]
flat_grid = grid.reshape(-1, data.shape[1])
# عنوان للمخططات
titles = [
"SVC with rbf kernel",
"SVC (linear kernel)\n with Fourier rbf feature map\nn_components=100",
"SVC (linear kernel)\n with Nystroem rbf feature map\nn_components=100",
]
plt.figure(figsize=(18, 7.5))
plt.rcParams.update({"font.size": 14})
# التنبؤ والرسم
for i, clf in enumerate((kernel_svm, nystroem_approx_svm, fourier_approx_svm)):
# رسم حدود القرار. لهذا، سنقوم بتعيين لون لكل نقطة
# في الشبكة [x_min, x_max]x[y_min, y_max].
plt.subplot(1, 3, i + 1)
Z = clf.predict(flat_grid)
# وضع النتيجة في رسم ملون
Z = Z.reshape(grid.shape[:-1])
levels = np.arange(10)
lv_eps = 0.01 # ضبط خريطة من مستويات التضاريس المحسوبة إلى لون.
plt.contourf(
multiples,
multiples,
Z,
levels=levels - lv_eps,
cmap=plt.cm.tab10,
vmin=0,
vmax=10,
alpha=0.7,
)
plt.axis("off")
# رسم نقاط التدريب أيضًا
plt.scatter(
X[:, 0],
X[:, 1],
c=targets_train,
cmap=plt.cm.tab10,
edgecolors=(0, 0, 0),
vmin=0,
vmax=10,
)
plt.title(titles[i])
plt.tight_layout()
plt.show()
Total running time of the script: (0 minutes 2.768 seconds)
Related examples
رسم مخططات لمصنفات SVM المختلفة في مجموعة بيانات الزهرة
حدود القرار للمصنفات شبه المُشرفة مقابل SVM على مجموعة بيانات Iris