ملاحظة
Go to the end to download the full example code. or to run this example in your browser via JupyterLite or Binder
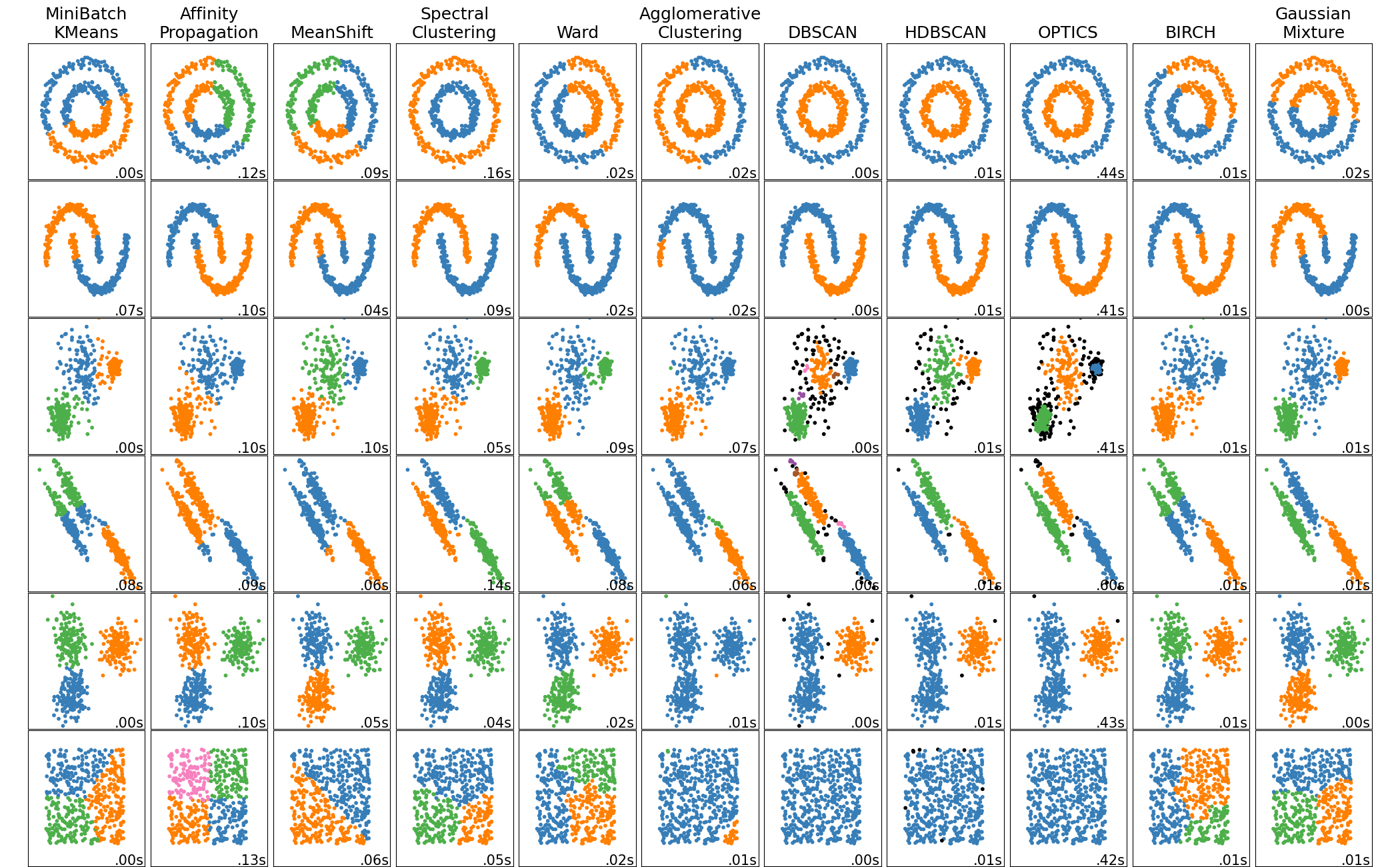
مقارنة خوارزميات التجميع المختلفة على مجموعات البيانات التجريبية#
هذا المثال يوضح خصائص خوارزميات التجميع المختلفة على مجموعات البيانات التي تعتبر "مثيرة للاهتمام" ولكنها لا تزال ثنائية الأبعاد. باستثناء مجموعة البيانات الأخيرة، تم ضبط معلمات كل من هذه الأزواج من مجموعة البيانات والخوارزمية لإنتاج نتائج تجميع جيدة. بعض الخوارزميات أكثر حساسية لقيم المعلمات من غيرها.
مجموعة البيانات الأخيرة هي مثال على حالة "عدم وجود" التجميع: البيانات متجانسة، ولا يوجد تجميع جيد. في هذا المثال، تستخدم مجموعة البيانات "العدمية" نفس المعلمات مثل مجموعة البيانات في الصف أعلاه، مما يمثل عدم تطابق في قيم المعلمات وبنية البيانات.
في حين أن هذه الأمثلة تعطي بعض الحدس حول الخوارزميات، قد لا ينطبق هذا الحدس على البيانات ذات الأبعاد العالية جدا.
# المؤلفون: مطوري scikit-learn
# معرف الترخيص: BSD-3-Clause
import time
import warnings
from itertools import cycle, islice
import matplotlib.pyplot as plt
import numpy as np
from sklearn import cluster, datasets, mixture
from sklearn.neighbors import kneighbors_graph
from sklearn.preprocessing import StandardScaler
# ============
# توليد مجموعات البيانات. نختار الحجم كبير بما يكفي لرؤية قابلية التوسع
# للخوارزميات، ولكن ليس كبيرا جدا لتجنب أوقات التشغيل الطويلة
# ============
n_samples = 500
seed = 30
noisy_circles = datasets.make_circles(
n_samples=n_samples, factor=0.5, noise=0.05, random_state=seed
)
noisy_moons = datasets.make_moons(
n_samples=n_samples, noise=0.05, random_state=seed)
blobs = datasets.make_blobs(n_samples=n_samples, random_state=seed)
rng = np.random.RandomState(seed)
no_structure = rng.rand(n_samples, 2), None
# البيانات الموزعة بشكل غير متساو
random_state = 170
X, y = datasets.make_blobs(n_samples=n_samples, random_state=random_state)
transformation = [[0.6, -0.6], [-0.4, 0.8]]
X_aniso = np.dot(X, transformation)
aniso = (X_aniso, y)
# blobs مع تباين متغير
varied = datasets.make_blobs(
n_samples=n_samples, cluster_std=[1.0, 2.5, 0.5], random_state=random_state
)
# ============
# ضبط معلمات التجميع
# ============
plt.figure(figsize=(9 * 2 + 3, 13))
plt.subplots_adjust(
left=0.02, right=0.98, bottom=0.001, top=0.95, wspace=0.05, hspace=0.01
)
plot_num = 1
default_base = {
"quantile": 0.3,
"eps": 0.3,
"damping": 0.9,
"preference": -200,
"n_neighbors": 3,
"n_clusters": 3,
"min_samples": 7,
"xi": 0.05,
"min_cluster_size": 0.1,
"allow_single_cluster": True,
"hdbscan_min_cluster_size": 15,
"hdbscan_min_samples": 3,
"random_state": 42,
}
datasets = [
(
noisy_circles,
{
"damping": 0.77,
"preference": -240,
"quantile": 0.2,
"n_clusters": 2,
"min_samples": 7,
"xi": 0.08,
},
),
(
noisy_moons,
{
"damping": 0.75,
"preference": -220,
"n_clusters": 2,
"min_samples": 7,
"xi": 0.1,
},
),
(
varied,
{
"eps": 0.18,
"n_neighbors": 2,
"min_samples": 7,
"xi": 0.01,
"min_cluster_size": 0.2,
},
),
(
aniso,
{
"eps": 0.15,
"n_neighbors": 2,
"min_samples": 7,
"xi": 0.1,
"min_cluster_size": 0.2,
},
),
(blobs, {"min_samples": 7, "xi": 0.1, "min_cluster_size": 0.2}),
(no_structure, {}),
]
for i_dataset, (dataset, algo_params) in enumerate(datasets):
# تحديث المعلمات بقيم خاصة بمجموعة البيانات
params = default_base.copy()
params.update(algo_params)
X, y = dataset
# تطبيع مجموعة البيانات لسهولة اختيار المعلمات
X = StandardScaler().fit_transform(X)
# تقدير عرض النطاق الترددي لمتوسط التحول
bandwidth = cluster.estimate_bandwidth(X, quantile=params["quantile"])
# تقدير عرض النطاق الترددي لمتوسط التحول
bandwidth = cluster.estimate_bandwidth(X, quantile=params["quantile"])
# مصفوفة الاتصال للتجمع الهيكلي
connectivity = kneighbors_graph(
X, n_neighbors=params["n_neighbors"], include_self=False
)
# جعل الاتصال متماثل
connectivity = 0.5 * (connectivity + connectivity.T)
# ============
# إنشاء كائنات التجميع
# ============
ms = cluster.MeanShift(bandwidth=bandwidth, bin_seeding=True)
two_means = cluster.MiniBatchKMeans(
n_clusters=params["n_clusters"],
random_state=params["random_state"],
)
ward = cluster.AgglomerativeClustering(
n_clusters=params["n_clusters"], linkage="ward", connectivity=connectivity
)
spectral = cluster.SpectralClustering(
n_clusters=params["n_clusters"],
eigen_solver="arpack",
affinity="nearest_neighbors",
random_state=params["random_state"],
)
dbscan = cluster.DBSCAN(eps=params["eps"])
hdbscan = cluster.HDBSCAN(
min_samples=params["hdbscan_min_samples"],
min_cluster_size=params["hdbscan_min_cluster_size"],
allow_single_cluster=params["allow_single_cluster"],
)
optics = cluster.OPTICS(
min_samples=params["min_samples"],
xi=params["xi"],
min_cluster_size=params["min_cluster_size"],
)
affinity_propagation = cluster.AffinityPropagation(
damping=params["damping"],
preference=params["preference"],
random_state=params["random_state"],
)
average_linkage = cluster.AgglomerativeClustering(
linkage="average",
metric="cityblock",
n_clusters=params["n_clusters"],
connectivity=connectivity,
)
birch = cluster.Birch(n_clusters=params["n_clusters"])
gmm = mixture.GaussianMixture(
n_components=params["n_clusters"],
covariance_type="full",
random_state=params["random_state"],
)
clustering_algorithms = (
("MiniBatch\nKMeans", two_means),
("Affinity\nPropagation", affinity_propagation),
("MeanShift", ms),
("Spectral\nClustering", spectral),
("Ward", ward),
("Agglomerative\nClustering", average_linkage),
("DBSCAN", dbscan),
("HDBSCAN", hdbscan),
("OPTICS", optics),
("BIRCH", birch),
("Gaussian\nMixture", gmm),
)
for name, algorithm in clustering_algorithms:
t0 = time.time()
# التقاط التحذيرات المتعلقة بـ kneighbors_graph
with warnings.catch_warnings():
warnings.filterwarnings(
"ignore",
message="the number of connected components of the "
+ "connectivity matrix is [0-9]{1,2}"
+ " > 1. Completing it to avoid stopping the tree early.",
category=UserWarning,
)
warnings.filterwarnings(
"ignore",
message="Graph is not fully connected, spectral embedding"
+ " may not work as expected.",
category=UserWarning,
)
algorithm.fit(X)
t1 = time.time()
if hasattr(algorithm, "labels_"):
y_pred = algorithm.labels_.astype(int)
else:
y_pred = algorithm.predict(X)
plt.subplot(len(datasets), len(clustering_algorithms), plot_num)
if i_dataset == 0:
plt.title(name, size=18)
colors = np.array(
list(
islice(
cycle(
[
"#377eb8",
"#ff7f00",
"#4daf4a",
"#f781bf",
"#a65628",
"#984ea3",
"#999999",
"#e41a1c",
"#dede00",
]
),
int(max(y_pred) + 1),
)
)
)
# إضافة اللون الأسود للبيانات الشاذة (إن وجدت)
colors = np.append(colors, ["#000000"])
plt.scatter(X[:, 0], X[:, 1], s=10, color=colors[y_pred])
plt.xlim(-2.5, 2.5)
plt.ylim(-2.5, 2.5)
plt.xticks(())
plt.yticks(())
plt.text(
0.99,
0.01,
("%.2fs" % (t1 - t0)).lstrip("0"),
transform=plt.gca().transAxes,
size=15,
horizontalalignment="right",
)
plot_num += 1
plt.show()
Total running time of the script: (0 minutes 7.120 seconds)
Related examples
مقارنة طرق الربط الهرمي المختلفة على مجموعات بيانات تجريبية